| #1: タンパク質 | UDP-3-O-(3-hydroxymyristoyl)glucosamine N-acyltransferase / UDP-3-O-(3-OHC14)-GlcN N-acyltransferase / UDP-3-O-(3-hydroxytetradecanoyl)glucosamine N-acyltransferase
分子量: 36101.305 Da / 分子数: 3 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌)遺伝子: lpxD, lpxD_1, lpxD_2, A8C65_17800, A9R57_04450, AC789_1c01770, ACN002_0184, ACN77_17005, ACN81_28485, ACU57_18525, ACU90_26495, AM270_18210, AML07_22885, AML35_22825, APZ14_11060, AUQ13_ ...遺伝子: lpxD, lpxD_1, lpxD_2, A8C65_17800, A9R57_04450, AC789_1c01770, ACN002_0184, ACN77_17005, ACN81_28485, ACU57_18525, ACU90_26495, AM270_18210, AML07_22885, AML35_22825, APZ14_11060, AUQ13_07450, AUS26_23195, AW059_09890, AW106_08805, B1K96_12940, B7C53_01310, BANRA_00558, BANRA_00653, BANRA_02262, BANRA_03350, BET08_19125, BHF46_07835, BHS81_00700, BHS87_00855, BIZ41_19025, BJJ90_21310, BK292_13720, BK400_03560, BMT53_09465, BN17_46021, BTQ06_06540, BUE81_01655, BVL39_02135, BW690_02415, BZL31_14885, C2U48_09555, C4J69_03380, C5N07_08630, C5P01_08975, C5P43_07335, C6669_04195, C6986_01145, C7235_20180, C7B06_05915, C7B07_04225, CA593_02815, CG691_14440, CG692_09445, CG705_12975, CG706_12690, COD30_14715, COD46_06685, CR538_20590, CR539_04985, CRM83_15040, CXB56_23080, D0X26_09365, D2184_08545, D2185_20180, D2F89_01460, D3821_06260, D3O91_03240, D3Y67_16455, D9D20_18680, D9D55_03030, D9D69_06830, D9E22_13300, D9E35_14480, D9G42_07535, D9H66_18155, D9H68_13825, D9I18_15010, D9I87_01590, D9I97_03755, D9J11_16875, D9J44_14030, DIV22_06320, DL545_20340, DMZ31_06810, DNQ41_04690, DNQ45_10065, DQF57_05615, DQO13_06200, DS732_05825, DTL43_05870, EAI42_08350, EAI46_03595, EAI52_11055, EC1094V2_3672, EC3234A_2c01610, EC3426_00931, EC382_03005, ECTO6_03883, ED287_04550, ED600_04595, EEP23_00115, EFV01_09910, EFV02_20465, EFV04_15215, EFV08_17150, EFV11_08135, EFV12_18845, EFV15_17225, EFV17_09875, EIA13_13600, EL75_3585, EL79_3695, EL80_3642, ERS085374_03841, ERS150873_01122, ERS150876_00054, FORC28_5013, HW43_04650, JD73_13090, NCTC10429_04183, NCTC10865_05054, NCTC11022_04906, NCTC11126_03647, NCTC13462_02218, NCTC8500_04533, NCTC8960_01551, NCTC9037_04178, NCTC9045_04699, NCTC9055_00956, NCTC9058_02808, NCTC9062_04225, NCTC9706_01326, NCTC9969_04255, PU06_22990, RG28_03860, RK56_027945, RX35_04913, SAMEA3472043_02537, SAMEA3472044_04146, SAMEA3472047_02509, SAMEA3472070_02384, SAMEA3472080_03763, SAMEA3472114_03609, SAMEA3484427_01386, SAMEA3484429_01095, SAMEA3752553_02884, SAMEA3752557_00885, SAMEA3752559_03701, SAMEA3753064_03381, SAMEA3753097_03916, SAMEA3753290_00404, SAMEA3753300_01852, SK85_00179, WQ89_09605, WR15_02380プラスミド Escherichia coli (大腸菌)遺伝子: lpxD, lpxD_1, lpxD_2, A8C65_17800, A9R57_04450, AC789_1c01770, ACN002_0184, ACN77_17005, ACN81_28485, ACU57_18525, ACU90_26495, AM270_18210, AML07_22885, AML35_22825, APZ14_11060, AUQ13_ ...遺伝子: lpxD, lpxD_1, lpxD_2, A8C65_17800, A9R57_04450, AC789_1c01770, ACN002_0184, ACN77_17005, ACN81_28485, ACU57_18525, ACU90_26495, AM270_18210, AML07_22885, AML35_22825, APZ14_11060, AUQ13_07450, AUS26_23195, AW059_09890, AW106_08805, B1K96_12940, B7C53_01310, BANRA_00558, BANRA_00653, BANRA_02262, BANRA_03350, BET08_19125, BHF46_07835, BHS81_00700, BHS87_00855, BIZ41_19025, BJJ90_21310, BK292_13720, BK400_03560, BMT53_09465, BN17_46021, BTQ06_06540, BUE81_01655, BVL39_02135, BW690_02415, BZL31_14885, C2U48_09555, C4J69_03380, C5N07_08630, C5P01_08975, C5P43_07335, C6669_04195, C6986_01145, C7235_20180, C7B06_05915, C7B07_04225, CA593_02815, CG691_14440, CG692_09445, CG705_12975, CG706_12690, COD30_14715, COD46_06685, CR538_20590, CR539_04985, CRM83_15040, CXB56_23080, D0X26_09365, D2184_08545, D2185_20180, D2F89_01460, D3821_06260, D3O91_03240, D3Y67_16455, D9D20_18680, D9D55_03030, D9D69_06830, D9E22_13300, D9E35_14480, D9G42_07535, D9H66_18155, D9H68_13825, D9I18_15010, D9I87_01590, D9I97_03755, D9J11_16875, D9J44_14030, DIV22_06320, DL545_20340, DMZ31_06810, DNQ41_04690, DNQ45_10065, DQF57_05615, DQO13_06200, DS732_05825, DTL43_05870, EAI42_08350, EAI46_03595, EAI52_11055, EC1094V2_3672, EC3234A_2c01610, EC3426_00931, EC382_03005, ECTO6_03883, ED287_04550, ED600_04595, EEP23_00115, EFV01_09910, EFV02_20465, EFV04_15215, EFV08_17150, EFV11_08135, EFV12_18845, EFV15_17225, EFV17_09875, EIA13_13600, EL75_3585, EL79_3695, EL80_3642, ERS085374_03841, ERS150873_01122, ERS150876_00054, FORC28_5013, HW43_04650, JD73_13090, NCTC10429_04183, NCTC10865_05054, NCTC11022_04906, NCTC11126_03647, NCTC13462_02218, NCTC8500_04533, NCTC8960_01551, NCTC9037_04178, NCTC9045_04699, NCTC9055_00956, NCTC9058_02808, NCTC9062_04225, NCTC9706_01326, NCTC9969_04255, PU06_22990, RG28_03860, RK56_027945, RX35_04913, SAMEA3472043_02537, SAMEA3472044_04146, SAMEA3472047_02509, SAMEA3472070_02384, SAMEA3472080_03763, SAMEA3472114_03609, SAMEA3484427_01386, SAMEA3484429_01095, SAMEA3752553_02884, SAMEA3752557_00885, SAMEA3752559_03701, SAMEA3753064_03381, SAMEA3753097_03916, SAMEA3753290_00404, SAMEA3753300_01852, SK85_00179, WQ89_09605, WR15_02380プラスミド: pET24a / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌)参照 Escherichia coli BL21(DE3) (大腸菌)参照: UniProt: Q0P6M7, UniProt: P21645*PLUS, UDP-3-O-(3-hydroxyacyl)glucosamine N-acyltransferase       | #2: 化合物 | ChemComp-MG / MAGNESIUM ION
  分子量 分子量: 24.305 Da / 分子数: 2 / 由来タイプ: 合成 / 式: Mg | #3: 化合物 | ChemComp-O3Y / 4-(2-chlorophenyl)-3-hydroxy-7,7-dimethyl-2-phenyl-2,6,7,8-tetrahydro-5H-pyrazolo[3,4-b]quinolin-5-one
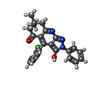  分子量 分子量: 417.888 Da / 分子数: 3 / 由来タイプ: 合成 / 式: C 24H 20ClN 3O 2 / タイプ: SUBJECT OF INVESTIGATION | #4: 水 | ChemComp-HOH / water
 分子量 分子量: 18.015 Da / 分子数: 981 / 由来タイプ: 天然 / 式: H 2O |
|---|
|
|---|
|
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.7 Å
分子置換 / 解像度: 1.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Acs Infect Dis. / 年: 2020
ジャーナル: Acs Infect Dis. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6p83.cif.gz
6p83.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6p83.ent.gz
pdb6p83.ent.gz PDB形式
PDB形式 6p83.json.gz
6p83.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6p83_validation.pdf.gz
6p83_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6p83_full_validation.pdf.gz
6p83_full_validation.pdf.gz 6p83_validation.xml.gz
6p83_validation.xml.gz 6p83_validation.cif.gz
6p83_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/p8/6p83
https://data.pdbj.org/pub/pdb/validation_reports/p8/6p83 ftp://data.pdbj.org/pub/pdb/validation_reports/p8/6p83
ftp://data.pdbj.org/pub/pdb/validation_reports/p8/6p83 リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.2 / 波長: 0.977408 Å
/ ビームライン: 5.0.2 / 波長: 0.977408 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj







