| 登録情報 | データベース: PDB / ID: 6o7g
|
|---|
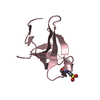
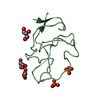
| タイトル | Solution structure of MLL4 PHD6 domain in complex with histone H4K16ac peptide |
|---|
 要素 要素 | - Histone H4
- Histone-lysine N-methyltransferase 2D
|
|---|
 キーワード キーワード | TRANSCRIPTION / MLL4 / PHD finger / H4K16ac / MOF / acetylation / histone / chromatin |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
beta-catenin-TCF complex assembly / oocyte growth / [histone H3]-lysine4 N-methyltransferase / histone H3K4 monomethyltransferase activity / histone H3K4 trimethyltransferase activity / Epigenetic regulation of gene expression by MLL3 and MLL4 complexes / MLL3/4 complex / histone H3K4 methyltransferase activity / oogenesis / positive regulation of intracellular estrogen receptor signaling pathway ...beta-catenin-TCF complex assembly / oocyte growth / [histone H3]-lysine4 N-methyltransferase / histone H3K4 monomethyltransferase activity / histone H3K4 trimethyltransferase activity / Epigenetic regulation of gene expression by MLL3 and MLL4 complexes / MLL3/4 complex / histone H3K4 methyltransferase activity / oogenesis / positive regulation of intracellular estrogen receptor signaling pathway / Formation of WDR5-containing histone-modifying complexes / Deactivation of the beta-catenin transactivating complex / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / PKMTs methylate histone lysines / response to estrogen / Activation of anterior HOX genes in hindbrain development during early embryogenesis / heterochromatin formation / MLL4 and MLL3 complexes regulate expression of PPARG target genes in adipogenesis and hepatic steatosis / methylation / transcription coactivator activity / transcription cis-regulatory region binding / positive regulation of cell population proliferation / regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / DNA binding / zinc ion binding / nucleoplasm / nucleus類似検索 - 分子機能 Histone-lysine N-methyltransferase 2D / KMT2D, ePHD1 domain / KMT2D, ePHD2 domain / : / : / FY-rich, N-terminal / F/Y-rich N-terminus / PHD-like zinc-binding domain / FYR domain FYRN motif profile. / "FY-rich" domain, N-terminal region ...Histone-lysine N-methyltransferase 2D / KMT2D, ePHD1 domain / KMT2D, ePHD2 domain / : / : / FY-rich, N-terminal / F/Y-rich N-terminus / PHD-like zinc-binding domain / FYR domain FYRN motif profile. / "FY-rich" domain, N-terminal region / FY-rich, C-terminal / F/Y rich C-terminus / FYR domain FYRC motif profile. / "FY-rich" domain, C-terminal region / Cysteine-rich motif following a subset of SET domains / Post-SET domain / Post-SET domain profile. / high mobility group / High mobility group box domain / High mobility group box domain superfamily / PHD-zinc-finger like domain / Extended PHD (ePHD) domain / Extended PHD (ePHD) domain profile. / SET (Su(var)3-9, Enhancer-of-zeste, Trithorax) domain / SET domain / SET domain superfamily / SET domain profile. / SET domain / PHD-finger / Zinc finger PHD-type signature. / Ring finger / Zinc finger PHD-type profile. / Zinc finger, PHD-finger / Zinc finger, PHD-type / PHD zinc finger / Zinc finger, FYVE/PHD-type / Zinc finger RING-type profile. / Zinc finger, RING-type / Zinc finger, RING/FYVE/PHD-type類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Zhang, Y. / Kutateladze, T.G. |
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019
タイトル: Selective binding of the PHD6 finger of MLL4 to histone H4K16ac links MLL4 and MOF.
著者: Zhang, Y. / Jang, Y. / Lee, J.E. / Ahn, J. / Xu, L. / Holden, M.R. / Cornett, E.M. / Krajewski, K. / Klein, B.J. / Wang, S.P. / Dou, Y. / Roeder, R.G. / Strahl, B.D. / Rothbart, S.B. / Shi, X. ...著者: Zhang, Y. / Jang, Y. / Lee, J.E. / Ahn, J. / Xu, L. / Holden, M.R. / Cornett, E.M. / Krajewski, K. / Klein, B.J. / Wang, S.P. / Dou, Y. / Roeder, R.G. / Strahl, B.D. / Rothbart, S.B. / Shi, X. / Ge, K. / Kutateladze, T.G. |
|---|
| 履歴 | | 登録 | 2019年3月7日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2019年5月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年6月12日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.identifier_ORCID / _citation_author.name |
|---|
| 改定 2.0 | 2023年11月15日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Database references / Derived calculations
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_remark / struct_conn / struct_conn_type
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_remark.text / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn_type.id |
|---|
| 改定 2.1 | 2024年11月13日 | Group: Database references / Structure summary
カテゴリ: database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI |
|---|
|
|---|
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR |
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Nat Commun / 年: 2019
ジャーナル: Nat Commun / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6o7g.cif.gz
6o7g.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6o7g.ent.gz
pdb6o7g.ent.gz PDB形式
PDB形式 6o7g.json.gz
6o7g.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6o7g_validation.pdf.gz
6o7g_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6o7g_full_validation.pdf.gz
6o7g_full_validation.pdf.gz 6o7g_validation.xml.gz
6o7g_validation.xml.gz 6o7g_validation.cif.gz
6o7g_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/o7/6o7g
https://data.pdbj.org/pub/pdb/validation_reports/o7/6o7g ftp://data.pdbj.org/pub/pdb/validation_reports/o7/6o7g
ftp://data.pdbj.org/pub/pdb/validation_reports/o7/6o7g リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: KMT2D, ALR, MLL2, MLL4 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: KMT2D, ALR, MLL2, MLL4 / 発現宿主: 
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj






 HNCA
HNCA