登録情報 データベース : PDB / ID : 6n68タイトル NMR solution structure of Protonectin (Agelaia pallipes pallipes) interacting with SDS micelles: an antimicrobial peptide with anticancer activity on breast cancer cells Protonectin キーワード / / / 機能・相同性 分子機能 構成要素
/ / / / / / / / / 生物種 Agelaia pallipes pallipes (昆虫)手法 / データ登録者 Fadel, V. / Martins, D.B. / Dos Santos Cabrera, M.P. 資金援助 組織 認可番号 国 Sao Paulo Research Foundation (FAPESP) 2012/24259-0
ジャーナル : J Colloid Interface Sci / 年 : 2021タイトル : Protonectin peptides target lipids, act at the interface and selectively kill metastatic breast cancer cells while preserving morphological integrity.著者 : Batista Martins, D. / Fadel, V. / Oliveira, F.D. / Gaspar, D. / Alvares, D.S. / Castanho, M.A.R.B. / Dos Santos Cabrera, M.P. 履歴 登録 2018年11月26日 登録サイト / 処理サイト 改定 1.0 2020年6月3日 Provider / タイプ 改定 1.1 2021年6月16日 Group / カテゴリ / citation_authorItem _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year 改定 1.2 2023年6月14日 Group / Other / カテゴリ / pdbx_database_statusItem / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Agelaia pallipes pallipes (昆虫)
Agelaia pallipes pallipes (昆虫) データ登録者
データ登録者 ブラジル, 1件
ブラジル, 1件  引用
引用 ジャーナル: J Colloid Interface Sci / 年: 2021
ジャーナル: J Colloid Interface Sci / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6n68.cif.gz
6n68.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6n68.ent.gz
pdb6n68.ent.gz PDB形式
PDB形式 6n68.json.gz
6n68.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6n68_validation.pdf.gz
6n68_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6n68_full_validation.pdf.gz
6n68_full_validation.pdf.gz 6n68_validation.xml.gz
6n68_validation.xml.gz 6n68_validation.cif.gz
6n68_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n6/6n68
https://data.pdbj.org/pub/pdb/validation_reports/n6/6n68 ftp://data.pdbj.org/pub/pdb/validation_reports/n6/6n68
ftp://data.pdbj.org/pub/pdb/validation_reports/n6/6n68 リンク
リンク 集合体
集合体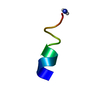
 要素
要素 Agelaia pallipes pallipes (昆虫) / 参照: UniProt: P69437
Agelaia pallipes pallipes (昆虫) / 参照: UniProt: P69437 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj GROMACS
GROMACS