+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6jgr | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of barley exohydrolaseI W434Y in complex with 4'-nitrophenyl thiolaminaribioside | ||||||
 要素 要素 | BETA-D-GLUCAN GLUCOHYDROLASE ISOENZYME EXO1 | ||||||
 キーワード キーワード | HYDROLASE / Barley exohydrolaseI / enzyme function | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane => GO:0016020 / hydrolase activity, hydrolyzing O-glycosyl compounds / carbohydrate metabolic process 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.25 Å 分子置換 / 解像度: 2.25 Å | ||||||
 データ登録者 データ登録者 | Luang, S. / Streltsov, V.A. / Hrmova, M. | ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: The evolutionary advantage of an aromatic clamp in plant family 3 glycoside exo-hydrolases. 著者: Luang, S. / Fernandez-Luengo, X. / Nin-Hill, A. / Streltsov, V.A. / Schwerdt, J.G. / Alonso-Gil, S. / Ketudat Cairns, J.R. / Pradeau, S. / Fort, S. / Marechal, J.D. / Masgrau, L. / Rovira, C. / Hrmova, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6jgr.cif.gz 6jgr.cif.gz | 253.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6jgr.ent.gz pdb6jgr.ent.gz | 199.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6jgr.json.gz 6jgr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6jgr_validation.pdf.gz 6jgr_validation.pdf.gz | 838.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6jgr_full_validation.pdf.gz 6jgr_full_validation.pdf.gz | 859.8 KB | 表示 | |
| XML形式データ |  6jgr_validation.xml.gz 6jgr_validation.xml.gz | 30.3 KB | 表示 | |
| CIF形式データ |  6jgr_validation.cif.gz 6jgr_validation.cif.gz | 43 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jg/6jgr https://data.pdbj.org/pub/pdb/validation_reports/jg/6jgr ftp://data.pdbj.org/pub/pdb/validation_reports/jg/6jgr ftp://data.pdbj.org/pub/pdb/validation_reports/jg/6jgr | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6jg1C  6jg2C  6jg6C  6jg7C  6jgaC  6jgbC  6jgcC  6jgdC  6jgeC  6jggC  6jgkC  6jglC  6jgnC  6jgoC  6jgpC  6jgqC  6jgsC  6jgtC  6k6vC  6kufC  6l1jC  6lbbC  6lbvC  6lc5C  3wliS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 65871.031 Da / 分子数: 1 / 変異: W434Y / 由来タイプ: 組換発現 由来: (組換発現)  発現宿主:  Komagataella pastoris (菌類) / 参照: UniProt: A0A287SCR5, UniProt: Q9XEI3*PLUS Komagataella pastoris (菌類) / 参照: UniProt: A0A287SCR5, UniProt: Q9XEI3*PLUS |
|---|
-非ポリマー , 5種, 334分子 
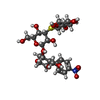







| #2: 化合物 | | #3: 化合物 | ChemComp-LAM / | #4: 化合物 | ChemComp-1PE / #5: 化合物 | ChemComp-ACT / #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|---|
| 配列の詳細 | Lys320 is confirmed by DNA sequencing result. However, the electron density map is not clear, ...Lys320 is confirmed by DNA sequencing result. However, the electron density map is not clear, probably side chain of this residue is flexible. |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.44 Å3/Da / 溶媒含有率: 64.29 % |
|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7 詳細: 1.7 M ammonium sulfate, 75 mM HEPES-NaOH buffer, pH 7, containing 7.5 mM sodium acetate and 1.2% (w/v) PEG 400 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Australian Synchrotron Australian Synchrotron  / ビームライン: MX1 / 波長: 0.9537 Å / ビームライン: MX1 / 波長: 0.9537 Å |
| 検出器 | タイプ: ADSC QUANTUM 210r / 検出器: CCD / 日付: 2012年11月22日 / 詳細: COLLIMATING MIRROR |
| 放射 | モノクロメーター: DOUBLE-CRYSTAL SI(111) MONOCHROMATOR プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9537 Å / 相対比: 1 |
| 反射 | 解像度: 2.25→87.71 Å / Num. obs: 42365 / % possible obs: 99.8 % / 冗長度: 29 % / Rmerge(I) obs: 0.158 / Net I/σ(I): 26.4 |
| 反射 シェル | 解像度: 2.25→2.3 Å / Rmerge(I) obs: 0.875 / Num. unique obs: 111351 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3WLI 解像度: 2.25→45.85 Å / Cor.coef. Fo:Fc: 0.955 / Cor.coef. Fo:Fc free: 0.934 / SU B: 8.289 / SU ML: 0.108 / 交差検証法: THROUGHOUT / ESU R: 0.178 / ESU R Free: 0.164 / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 32.965 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.25→45.85 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


