+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6hev | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal Structure of Ephrin A2 (EphA2) Receptor Protein Kinase with the NVP-BHG712 derivative AT061 | ||||||
 要素 要素 | Ephrin type-A receptor 2 | ||||||
 キーワード キーワード | TRANSFERASE / Inhibitor / Complex / Protein Tyrosine Kinase | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報notochord cell development / notochord formation / blood vessel endothelial cell proliferation involved in sprouting angiogenesis / negative regulation of lymphangiogenesis / lens fiber cell morphogenesis / axial mesoderm formation / cAMP metabolic process / regulation of blood vessel endothelial cell migration / pericyte cell differentiation / ephrin receptor activity ...notochord cell development / notochord formation / blood vessel endothelial cell proliferation involved in sprouting angiogenesis / negative regulation of lymphangiogenesis / lens fiber cell morphogenesis / axial mesoderm formation / cAMP metabolic process / regulation of blood vessel endothelial cell migration / pericyte cell differentiation / ephrin receptor activity / leading edge membrane / negative regulation of chemokine production / response to growth factor / post-anal tail morphogenesis / activation of GTPase activity / bone remodeling / positive regulation of bicellular tight junction assembly / regulation of lamellipodium assembly / branching involved in mammary gland duct morphogenesis / EPH-Ephrin signaling / central nervous system neuron differentiation / RND1 GTPase cycle / RND2 GTPase cycle / tight junction / RND3 GTPase cycle / neural tube development / mammary gland epithelial cell proliferation / RHOV GTPase cycle / growth factor binding / EPHA-mediated growth cone collapse / regulation of cell adhesion mediated by integrin / RHOU GTPase cycle / lamellipodium membrane / RHOG GTPase cycle / EPH-ephrin mediated repulsion of cells / RAC2 GTPase cycle / RAC3 GTPase cycle / regulation of angiogenesis / ephrin receptor signaling pathway / regulation of ERK1 and ERK2 cascade / vasculogenesis / keratinocyte differentiation / RAC1 GTPase cycle / transmembrane receptor protein tyrosine kinase activity / cell surface receptor protein tyrosine kinase signaling pathway / osteoclast differentiation / molecular function activator activity / negative regulation of angiogenesis / skeletal system development / protein localization to plasma membrane / cell chemotaxis / positive regulation of protein localization to plasma membrane / cell motility / receptor protein-tyrosine kinase / ruffle membrane / intrinsic apoptotic signaling pathway in response to DNA damage / osteoblast differentiation / cell migration / lamellipodium / virus receptor activity / angiogenesis / receptor complex / cell adhesion / defense response to Gram-positive bacterium / positive regulation of cell migration / cadherin binding / inflammatory response / focal adhesion / cell surface / ATP binding / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.28 Å 分子置換 / 解像度: 1.28 Å | ||||||
 データ登録者 データ登録者 | Kudlinzki, D. / Troester, A. / Witt, K. / Linhard, V.L. / Gande, S.L. / Saxena, K. / Schwalbe, H. | ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Effects of NVP-BHG712 chemical modifications on EPHA2 binding and affinity 著者: Troester, A. / Kudlinzki, D. / Saxena, K. / Gande, S. / Schwalbe, H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6hev.cif.gz 6hev.cif.gz | 133.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6hev.ent.gz pdb6hev.ent.gz | 102.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6hev.json.gz 6hev.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6hev_validation.pdf.gz 6hev_validation.pdf.gz | 748.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6hev_full_validation.pdf.gz 6hev_full_validation.pdf.gz | 751.3 KB | 表示 | |
| XML形式データ |  6hev_validation.xml.gz 6hev_validation.xml.gz | 15.2 KB | 表示 | |
| CIF形式データ |  6hev_validation.cif.gz 6hev_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/he/6hev https://data.pdbj.org/pub/pdb/validation_reports/he/6hev ftp://data.pdbj.org/pub/pdb/validation_reports/he/6hev ftp://data.pdbj.org/pub/pdb/validation_reports/he/6hev | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6hesC  6hetC  6heuC  6hewC  6hexC  6heyC  6q7bC  6q7cC  6q7dC  6q7eC  6q7fC  6q7gC  6fnfS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 34462.840 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: EPHA2, ECK Homo sapiens (ヒト) / 遺伝子: EPHA2, ECK発現宿主:  参照: UniProt: P29317 |
|---|---|
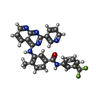
| #2: 化合物 | ChemComp-G0K / |
| #3: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.96 Å3/Da / 溶媒含有率: 37.14 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 37.5 % MPD/PEG1000/PEG3350 (MD), 0.1 M Amino Acids Mix (MD), 0.1 M Tris pH 7.5 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  PETRA III, EMBL c/o DESY PETRA III, EMBL c/o DESY  / ビームライン: P13 (MX1) / 波長: 1.03318 Å / ビームライン: P13 (MX1) / 波長: 1.03318 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M-F / 検出器: PIXEL / 日付: 2016年9月15日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.03318 Å / 相対比: 1 |
| 反射 | 解像度: 1.28→38.4 Å / Num. obs: 65628 / % possible obs: 96.4 % / 冗長度: 6.2 % / Biso Wilson estimate: 20.85 Å2 / CC1/2: 0.997 / Rrim(I) all: 0.107 / Net I/σ(I): 9.34 |
| 反射 シェル | 解像度: 1.28→1.36 Å / 冗長度: 4.42 % / Mean I/σ(I) obs: 1.11 / Num. unique obs: 8923 / CC1/2: 0.201 / % possible all: 81.3 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 6FNF 解像度: 1.28→31.023 Å / SU ML: 0.21 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 24.65
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.28→31.023 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj