+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6e7t | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Heterodimer of the GluN1b-GluN2B NMDA receptor amino-terminal domains bound to allosteric inhibitor 93-6 | |||||||||
 要素 要素 | (Glutamate receptor ionotropic, NMDA ...) x 2 | |||||||||
 キーワード キーワード | TRANSPORT PROTEIN / NMDA Receptor / Ion channel / Allosteric modulation / Extracellular domain | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to curcumin / cellular response to corticosterone stimulus / cellular response to magnesium starvation / sensory organ development / sensitization / EPHB-mediated forward signaling / Assembly and cell surface presentation of NMDA receptors / response to hydrogen sulfide / auditory behavior / dendritic branch ...cellular response to curcumin / cellular response to corticosterone stimulus / cellular response to magnesium starvation / sensory organ development / sensitization / EPHB-mediated forward signaling / Assembly and cell surface presentation of NMDA receptors / response to hydrogen sulfide / auditory behavior / dendritic branch / response to other organism / regulation of ARF protein signal transduction / fear response / apical dendrite / positive regulation of inhibitory postsynaptic potential / suckling behavior / response to methylmercury / response to manganese ion / response to carbohydrate / interleukin-1 receptor binding / cellular response to dsRNA / response to growth hormone / cellular response to lipid / negative regulation of dendritic spine maintenance / heterocyclic compound binding / positive regulation of glutamate secretion / regulation of monoatomic cation transmembrane transport / RAF/MAP kinase cascade / NMDA glutamate receptor activity / Synaptic adhesion-like molecules / response to glycoside / NMDA selective glutamate receptor complex / glutamate binding / ligand-gated sodium channel activity / response to zinc ion / calcium ion transmembrane import into cytosol / protein heterotetramerization / glycine binding / response to amine / regulation of cAMP/PKA signal transduction / small molecule binding / parallel fiber to Purkinje cell synapse / receptor clustering / startle response / monoatomic cation transmembrane transport / behavioral response to pain / regulation of MAPK cascade / regulation of postsynaptic membrane potential / response to magnesium ion / associative learning / action potential / extracellularly glutamate-gated ion channel activity / response to electrical stimulus / monoatomic cation transport / regulation of neuronal synaptic plasticity / glutamate receptor binding / Unblocking of NMDA receptors, glutamate binding and activation / positive regulation of excitatory postsynaptic potential / long-term memory / detection of mechanical stimulus involved in sensory perception of pain / response to mechanical stimulus / synaptic cleft / neuron development / behavioral fear response / multicellular organismal response to stress / positive regulation of synaptic transmission, glutamatergic / postsynaptic density, intracellular component / monoatomic cation channel activity / response to fungicide / glutamate-gated receptor activity / cell adhesion molecule binding / regulation of long-term synaptic depression / D2 dopamine receptor binding / cellular response to manganese ion / presynaptic active zone membrane / glutamate-gated calcium ion channel activity / response to cytokine / ionotropic glutamate receptor binding / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / cellular response to forskolin / sodium ion transmembrane transport / ionotropic glutamate receptor signaling pathway / protein tyrosine kinase binding / synaptic membrane / response to amphetamine / hippocampal mossy fiber to CA3 synapse / learning / response to nicotine / regulation of membrane potential / excitatory postsynaptic potential / response to cocaine / hippocampus development / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / synaptic transmission, glutamatergic / cellular response to amino acid stimulus / regulation of long-term neuronal synaptic plasticity / response to calcium ion / postsynaptic density membrane / cerebral cortex development / beta-catenin binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.31 Å 分子置換 / 解像度: 2.31 Å | |||||||||
 データ登録者 データ登録者 | Regan, M.C. / Furukawa, H. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Structural elements of a pH-sensitive inhibitor binding site in NMDA receptors 著者: Regan, M.C. / Furukawa, H. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6e7t.cif.gz 6e7t.cif.gz | 568 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6e7t.ent.gz pdb6e7t.ent.gz | 464.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6e7t.json.gz 6e7t.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6e7t_validation.pdf.gz 6e7t_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6e7t_full_validation.pdf.gz 6e7t_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  6e7t_validation.xml.gz 6e7t_validation.xml.gz | 54.5 KB | 表示 | |
| CIF形式データ |  6e7t_validation.cif.gz 6e7t_validation.cif.gz | 74.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/e7/6e7t https://data.pdbj.org/pub/pdb/validation_reports/e7/6e7t ftp://data.pdbj.org/pub/pdb/validation_reports/e7/6e7t ftp://data.pdbj.org/pub/pdb/validation_reports/e7/6e7t | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Refine code: 1
NCSアンサンブル:
NCS oper:
|
- 要素
要素
-Glutamate receptor ionotropic, NMDA ... , 2種, 4分子 ACBD
| #1: タンパク質 | 分子量: 43162.270 Da / 分子数: 2 / 断片: Extracellular residues 23-407 / 変異: N61Q, N371Q / 由来タイプ: 組換発現 由来: (組換発現) 遺伝子: grin1 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: A0A1L8F5J9 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: A0A1L8F5J9#2: タンパク質 | 分子量: 41280.820 Da / 分子数: 2 / 断片: Extracellular residues 32-394 / 変異: N348D / 由来タイプ: 組換発現 / 由来: (組換発現)   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q00960 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q00960 |
|---|
-糖 , 2種, 7分子 
| #3: 多糖 | alpha-D-mannopyranose-(1-3)-[alpha-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-4)-2-acetamido-2- ...alpha-D-mannopyranose-(1-3)-[alpha-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |
|---|---|
| #4: 糖 | ChemComp-NAG / |
-非ポリマー , 4種, 332分子 

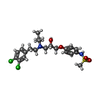




| #5: 化合物 | | #6: 化合物 | ChemComp-CL / #7: 化合物 | #8: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.27 Å3/Da / 溶媒含有率: 62.4 % |
|---|---|
| 結晶化 | 温度: 290 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 3.0-3.5 M sodium formate, 0.1 M HEPES, 35 mM sodium chloride, 7 mM Tris-HCl, 50 uM Ifenprodil |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS-II NSLS-II  / ビームライン: 17-ID-2 / 波長: 0.91 Å / ビームライン: 17-ID-2 / 波長: 0.91 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2018年2月2日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 0.91 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.31→50 Å / Num. obs: 83106 / % possible obs: 91 % / 冗長度: 3.6 % / Rmerge(I) obs: 0.092 / Rpim(I) all: 0.054 / Rrim(I) all: 0.107 / Χ2: 0.923 / Net I/σ(I): 7.7 / Num. measured all: 299032 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3QEL 解像度: 2.31→25 Å / Cor.coef. Fo:Fc: 0.942 / Cor.coef. Fo:Fc free: 0.921 / SU B: 15.019 / SU ML: 0.173 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.351 / ESU R Free: 0.235 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : WITH TLS ADDED
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 146.95 Å2 / Biso mean: 42.339 Å2 / Biso min: 14.13 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.31→25 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Dom-ID: 1 / Refine-ID: X-RAY DIFFRACTION / タイプ: TIGHT THERMAL / Weight position: 0.5
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.311→2.37 Å / Rfactor Rfree error: 0 / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj







