+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6dst | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Recombinant melittin | |||||||||
 要素 要素 | Melittin | |||||||||
 キーワード キーワード | TOXIN / Hemolytic / Antibacterial / Alpha-helical peptide / Bee venom | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報other organism cell membrane / molecular function inhibitor activity / porin activity / pore complex / protein kinase inhibitor activity / monoatomic ion transport / toxin activity / killing of cells of another organism / lipid binding / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 溶液NMR / simulated annealing | |||||||||
 データ登録者 データ登録者 | Ramirez, L.M. / Pande, J. / Shekhtman, A. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Phys Chem B / 年: 2019 ジャーナル: J Phys Chem B / 年: 2019タイトル: Helical Structure of Recombinant Melittin. 著者: Ramirez, L.S. / Pande, J. / Shekhtman, A. #1: ジャーナル: Biochemistry / 年: 2018 タイトル: Nuclear Magnetic Resonance-Based Structural Characterization and Backbone Dynamics of Recombinant Bee Venom Melittin. 著者: Ramirez, L. / Shekhtman, A. / Pande, J. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6dst.cif.gz 6dst.cif.gz | 197.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6dst.ent.gz pdb6dst.ent.gz | 165.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6dst.json.gz 6dst.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6dst_validation.pdf.gz 6dst_validation.pdf.gz | 464.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6dst_full_validation.pdf.gz 6dst_full_validation.pdf.gz | 795.1 KB | 表示 | |
| XML形式データ |  6dst_validation.xml.gz 6dst_validation.xml.gz | 45.7 KB | 表示 | |
| CIF形式データ |  6dst_validation.cif.gz 6dst_validation.cif.gz | 38.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ds/6dst https://data.pdbj.org/pub/pdb/validation_reports/ds/6dst ftp://data.pdbj.org/pub/pdb/validation_reports/ds/6dst ftp://data.pdbj.org/pub/pdb/validation_reports/ds/6dst | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
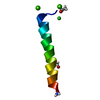
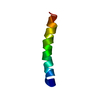
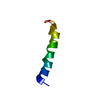
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 2852.487 Da / 分子数: 1 / 断片: residues 44-69 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MELT / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||
| 試料状態 |
|
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: Structures are based on 363 NOE-derived distance constraints, 252 dihedral angle restraints, and 24 lower-limit distance constraints | ||||||||||||||||||||
| 代表構造 | 選択基準: medoid | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj HNCA
HNCA