[English] 日本語
 Yorodumi
Yorodumi- PDB-6cqy: Crystal Structure of Recombinant Human Acetylcholinesterase in Co... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6cqy | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of Recombinant Human Acetylcholinesterase in Complex with EMPA and HI-6 | |||||||||
 Components Components | Acetylcholinesterase | |||||||||
 Keywords Keywords | HYDROLASE / Hydrolase Cholinesterase | |||||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of synaptic transmission, cholinergic / serine hydrolase activity / acetylcholine catabolic process in synaptic cleft / Neurotransmitter clearance / cholinesterase activity / acetylcholine catabolic process / acetylcholinesterase / amyloid precursor protein metabolic process / acetylcholine binding / osteoblast development ...negative regulation of synaptic transmission, cholinergic / serine hydrolase activity / acetylcholine catabolic process in synaptic cleft / Neurotransmitter clearance / cholinesterase activity / acetylcholine catabolic process / acetylcholinesterase / amyloid precursor protein metabolic process / acetylcholine binding / osteoblast development / acetylcholine receptor signaling pathway / acetylcholinesterase activity / Synthesis of PC / basement membrane / regulation of receptor recycling / Synthesis, secretion, and deacylation of Ghrelin / synaptic cleft / side of membrane / collagen binding / synapse assembly / laminin binding / positive regulation of protein secretion / neuromuscular junction / receptor internalization / nervous system development / amyloid-beta binding / positive regulation of cold-induced thermogenesis / retina development in camera-type eye / cell adhesion / hydrolase activity / synapse / perinuclear region of cytoplasm / cell surface / Golgi apparatus / protein homodimerization activity / extracellular space / extracellular region / nucleus / membrane / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.449 Å MOLECULAR REPLACEMENT / Resolution: 2.449 Å | |||||||||
 Authors Authors | Bester, S.M. / Guelta, M.A. / Pegan, S.D. / Height, J.J. | |||||||||
 Citation Citation |  Journal: Chem. Res. Toxicol. / Year: 2018 Journal: Chem. Res. Toxicol. / Year: 2018Title: Structural Insights of Stereospecific Inhibition of Human Acetylcholinesterase by VX and Subsequent Reactivation by HI-6. Authors: Bester, S.M. / Guelta, M.A. / Cheung, J. / Winemiller, M.D. / Bae, S.Y. / Myslinski, J. / Pegan, S.D. / Height, J.J. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6cqy.cif.gz 6cqy.cif.gz | 440.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6cqy.ent.gz pdb6cqy.ent.gz | 362.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6cqy.json.gz 6cqy.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/cq/6cqy https://data.pdbj.org/pub/pdb/validation_reports/cq/6cqy ftp://data.pdbj.org/pub/pdb/validation_reports/cq/6cqy ftp://data.pdbj.org/pub/pdb/validation_reports/cq/6cqy | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6cqtC  6cquC  6cqvC  6cqwC  6cqxC  6cqzC  4ey4S C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 59447.105 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: ACHE / Production host: Homo sapiens (human) / Gene: ACHE / Production host:  Homo sapiens (human) / References: UniProt: P22303, acetylcholinesterase Homo sapiens (human) / References: UniProt: P22303, acetylcholinesterase |
|---|
-Sugars , 2 types, 4 molecules 
| #2: Polysaccharide | Source method: isolated from a genetically manipulated source #3: Sugar | |
|---|
-Non-polymers , 3 types, 514 molecules 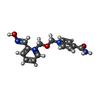
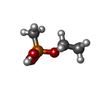



| #4: Chemical | | #5: Chemical | #6: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 4.35 Å3/Da / Density % sol: 71.75 % |
|---|---|
| Crystal grow | Temperature: 295 K / Method: vapor diffusion, sitting drop / pH: 7 Details: 15 to 21% PEG 3350, 0.17 - 0.21M potassium nitrate, pH 7.0, VAPOR DIFFUSION, SITTING DROP, temperature 295K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 22-ID / Wavelength: 1 Å / Beamline: 22-ID / Wavelength: 1 Å |
| Detector | Type: RAYONIX MX300-HS / Detector: CCD / Date: Jul 14, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 2.449→50 Å / Num. obs: 77545 / % possible obs: 100 % / Redundancy: 6.1 % / Rmerge(I) obs: 0.07 / Rpim(I) all: 0.031 / Net I/σ(I): 20 |
| Reflection shell | Resolution: 2.449→2.49 Å / Redundancy: 6.1 % / Rmerge(I) obs: 0.59 / Mean I/σ(I) obs: 2.1 / Num. unique obs: 7586 / Rpim(I) all: 0.275 / % possible all: 100 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4EY4 Resolution: 2.449→43.837 Å / SU ML: 0.22 / Cross valid method: THROUGHOUT / σ(F): 1.35 / Phase error: 21.2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.449→43.837 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller






















 PDBj
PDBj











