| 登録情報 | データベース: PDB / ID: 6bz3
|
|---|
| タイトル | Complex structure of FAK FAT domain and DCC P3 motif |
|---|
 要素 要素 | - Focal adhesion kinase 1
- Netrin receptor DCC
|
|---|
 キーワード キーワード | TRANSFERASE/MEMBRANE PROTEIN / 4-helix bundle / axon guidance / focal adhesion kinase / transferase-peptide complex / MEMBRANE PROTEIN / TRANSFERASE-MEMBRANE PROTEIN complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Netrin-1 signaling / DCC mediated attractive signaling / Caspase activation via Dependence Receptors in the absence of ligand / DCC mediated attractive signaling / Apoptotic cleavage of cellular proteins / netrin receptor activity / MET activates PTK2 signaling / dorsal/ventral axon guidance / NCAM signaling for neurite out-growth / negative regulation of synapse assembly ...Netrin-1 signaling / DCC mediated attractive signaling / Caspase activation via Dependence Receptors in the absence of ligand / DCC mediated attractive signaling / Apoptotic cleavage of cellular proteins / netrin receptor activity / MET activates PTK2 signaling / dorsal/ventral axon guidance / NCAM signaling for neurite out-growth / negative regulation of synapse assembly / spinal cord ventral commissure morphogenesis / anterior/posterior axon guidance / EPHB-mediated forward signaling / central nervous system neuron axonogenesis / RHO GTPases Activate WASPs and WAVEs / GRB2:SOS provides linkage to MAPK signaling for Integrins / Integrin signaling / p130Cas linkage to MAPK signaling for integrins / Turbulent (oscillatory, disturbed) flow shear stress activates signaling by PIEZO1 and integrins in endothelial cells / Extra-nuclear estrogen signaling / RAF/MAP kinase cascade / negative regulation of axonogenesis / growth cone membrane / negative regulation of organ growth / Regulation of actin dynamics for phagocytic cup formation / corpus callosum development / regulation of modification of postsynaptic actin cytoskeleton / VEGFA-VEGFR2 Pathway / optic nerve development / signal complex assembly / positive regulation of ubiquitin-dependent protein catabolic process / organ growth / nuclear migration / regulation of osteoblast differentiation / positive regulation of cardiac muscle hypertrophy / blood vessel development / axon development / regulation of cell adhesion mediated by integrin / positive regulation of protein kinase activity / ephrin receptor signaling pathway / endothelial cell migration / cellular response to transforming growth factor beta stimulus / postsynaptic modulation of chemical synaptic transmission / vasculogenesis / extracellular matrix organization / axonogenesis / negative regulation of autophagy / axon guidance / peptidyl-tyrosine phosphorylation / response to amphetamine / integrin-mediated signaling pathway / non-membrane spanning protein tyrosine kinase activity / non-specific protein-tyrosine kinase / cell-cell adhesion / positive regulation of neuron projection development / postsynaptic density membrane / cerebral cortex development / microtubule cytoskeleton organization / Schaffer collateral - CA1 synapse / positive regulation of protein phosphorylation / neuron migration / cell migration / regulation of cell shape / lamellipodium / regulation of cell population proliferation / protein autophosphorylation / protein tyrosine kinase activity / angiogenesis / cell cortex / transcription coactivator activity / positive regulation of ERK1 and ERK2 cascade / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / positive regulation of cell migration / ciliary basal body / apical plasma membrane / axon / focal adhesion / positive regulation of cell population proliferation / apoptotic process / centrosome / negative regulation of apoptotic process / perinuclear region of cytoplasm / glutamatergic synapse / cell surface / ATP binding / identical protein binding / nucleus / plasma membrane / cytosol類似検索 - 分子機能 Neogenin, C-terminal / Neogenin C-terminus / Nucleotidyltransferases domain 2 / Focal adhesion kinase, targeting (FAT) domain / Focal adhesion kinase, targeting (FAT) domain superfamily / Focal adhesion kinase, N-terminal / FAK1/PYK2, FERM domain C-lobe / Focal adhesion targeting region / FERM N-terminal domain / : ...Neogenin, C-terminal / Neogenin C-terminus / Nucleotidyltransferases domain 2 / Focal adhesion kinase, targeting (FAT) domain / Focal adhesion kinase, targeting (FAT) domain superfamily / Focal adhesion kinase, N-terminal / FAK1/PYK2, FERM domain C-lobe / Focal adhesion targeting region / FERM N-terminal domain / : / FAK1/PYK2, FERM domain C-lobe / FERM domain signature 2. / Immunoglobulin domain / FERM central domain / FERM/acyl-CoA-binding protein superfamily / FERM central domain / FERM superfamily, second domain / FERM domain / FERM domain profile. / Band 4.1 domain / Band 4.1 homologues / Immunoglobulin I-set / Immunoglobulin I-set domain / Fibronectin type III domain / Fibronectin type 3 domain / Four Helix Bundle (Hemerythrin (Met), subunit A) / Immunoglobulin subtype 2 / Immunoglobulin C-2 Type / Fibronectin type-III domain profile. / Fibronectin type III / Fibronectin type III superfamily / Tyrosine-protein kinase, catalytic domain / Tyrosine kinase, catalytic domain / Tyrosine protein kinases specific active-site signature. / PH-like domain superfamily / Immunoglobulin subtype / Immunoglobulin / Tyrosine-protein kinase, active site / Serine-threonine/tyrosine-protein kinase, catalytic domain / Protein tyrosine and serine/threonine kinase / Ubiquitin-like domain superfamily / Ig-like domain profile. / Immunoglobulin-like domain / Immunoglobulin-like domain superfamily / Protein kinase, ATP binding site / Protein kinases ATP-binding region signature. / Immunoglobulin-like fold / Protein kinase domain profile. / Protein kinase domain / Protein kinase-like domain superfamily / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 Focal adhesion kinase 1 / Netrin receptor DCC類似検索 - 構成要素 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ)
  Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.497 Å 分子置換 / 解像度: 2.497 Å |
|---|
 データ登録者 データ登録者 | Xu, S. / Wang, J.-H. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) | P01HL103526 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Cell Discov / 年: 2018 ジャーナル: Cell Discov / 年: 2018
タイトル: The binding of DCC-P3 motif and FAK-FAT domain mediates the initial step of netrin-1/DCC signaling for axon attraction.
著者: Xu, S. / Liu, Y. / Li, X. / Liu, Y. / Meijers, R. / Zhang, Y. / Wang, J.H. |
|---|
| 履歴 | | 登録 | 2017年12月22日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2018年4月4日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年12月4日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.2 | 2023年10月4日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.497 Å
分子置換 / 解像度: 2.497 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Cell Discov / 年: 2018
ジャーナル: Cell Discov / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6bz3.cif.gz
6bz3.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6bz3.ent.gz
pdb6bz3.ent.gz PDB形式
PDB形式 6bz3.json.gz
6bz3.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6bz3_validation.pdf.gz
6bz3_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6bz3_full_validation.pdf.gz
6bz3_full_validation.pdf.gz 6bz3_validation.xml.gz
6bz3_validation.xml.gz 6bz3_validation.cif.gz
6bz3_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/bz/6bz3
https://data.pdbj.org/pub/pdb/validation_reports/bz/6bz3 ftp://data.pdbj.org/pub/pdb/validation_reports/bz/6bz3
ftp://data.pdbj.org/pub/pdb/validation_reports/bz/6bz3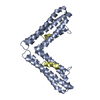
 リンク
リンク 集合体
集合体
 要素
要素


 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 19-ID / 波長: 0.979 Å
/ ビームライン: 19-ID / 波長: 0.979 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj















