+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6ba2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of MYST acetyltransferase domain in complex with inhibitor | |||||||||
 要素 要素 | Histone acetyltransferase KAT8 | |||||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  acetyltransferase / acetyltransferase /  inhibitor (酵素阻害剤) / inhibitor (酵素阻害剤) /  complex / MYST complex / MYST | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MSL complex / histone H4K16 acetyltransferase activity /  regulation of mRNA processing / myeloid cell differentiation / NSL complex / : / peptide-lysine-N-acetyltransferase activity / Formation of WDR5-containing histone-modifying complexes / regulation of mRNA processing / myeloid cell differentiation / NSL complex / : / peptide-lysine-N-acetyltransferase activity / Formation of WDR5-containing histone-modifying complexes /  MLL1 complex / MLL1 complex /  histone acetyltransferase complex ...MSL complex / histone H4K16 acetyltransferase activity / histone acetyltransferase complex ...MSL complex / histone H4K16 acetyltransferase activity /  regulation of mRNA processing / myeloid cell differentiation / NSL complex / : / peptide-lysine-N-acetyltransferase activity / Formation of WDR5-containing histone-modifying complexes / regulation of mRNA processing / myeloid cell differentiation / NSL complex / : / peptide-lysine-N-acetyltransferase activity / Formation of WDR5-containing histone-modifying complexes /  MLL1 complex / MLL1 complex /  histone acetyltransferase complex / histone acetyltransferase complex /  ヒストンアセチルトランスフェラーゼ / transcription initiation-coupled chromatin remodeling / ヒストンアセチルトランスフェラーゼ / transcription initiation-coupled chromatin remodeling /  神経発生 / 神経発生 /  regulation of autophagy / regulation of autophagy /  transcription coregulator activity / transcription coregulator activity /  動原体 / 動原体 /  nuclear matrix / HATs acetylate histones / RNA polymerase II-specific DNA-binding transcription factor binding / nuclear matrix / HATs acetylate histones / RNA polymerase II-specific DNA-binding transcription factor binding /  transcription coactivator activity / negative regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / transcription coactivator activity / negative regulation of DNA-templated transcription / positive regulation of DNA-templated transcription /  enzyme binding / positive regulation of transcription by RNA polymerase II / enzyme binding / positive regulation of transcription by RNA polymerase II /  核質 / 核質 /  metal ion binding / metal ion binding /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.85003821102 Å 分子置換 / 解像度: 1.85003821102 Å | |||||||||
 データ登録者 データ登録者 | Hermans, S.J. / Chung, M.C. / Peat, T.S. / Baell, J.B. / Thomas, T. / Parker, M.W. | |||||||||
| 資金援助 |  オーストラリア, 2件 オーストラリア, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. 著者: Baell, J.B. / Leaver, D.J. / Hermans, S.J. / Kelly, G.L. / Brennan, M.S. / Downer, N.L. / Nguyen, N. / Wichmann, J. / McRae, H.M. / Yang, Y. / Cleary, B. / Lagiakos, H.R. / Mieruszynski, S. / ...著者: Baell, J.B. / Leaver, D.J. / Hermans, S.J. / Kelly, G.L. / Brennan, M.S. / Downer, N.L. / Nguyen, N. / Wichmann, J. / McRae, H.M. / Yang, Y. / Cleary, B. / Lagiakos, H.R. / Mieruszynski, S. / Pacini, G. / Vanyai, H.K. / Bergamasco, M.I. / May, R.E. / Davey, B.K. / Morgan, K.J. / Sealey, A.J. / Wang, B. / Zamudio, N. / Wilcox, S. / Garnham, A.L. / Sheikh, B.N. / Aubrey, B.J. / Doggett, K. / Chung, M.C. / de Silva, M. / Bentley, J. / Pilling, P. / Hattarki, M. / Dolezal, O. / Dennis, M.L. / Falk, H. / Ren, B. / Charman, S.A. / White, K.L. / Rautela, J. / Newbold, A. / Hawkins, E.D. / Johnstone, R.W. / Huntington, N.D. / Peat, T.S. / Heath, J.K. / Strasser, A. / Parker, M.W. / Smyth, G.K. / Street, I.P. / Monahan, B.J. / Voss, A.K. / Thomas, T. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6ba2.cif.gz 6ba2.cif.gz | 86.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6ba2.ent.gz pdb6ba2.ent.gz | 55.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6ba2.json.gz 6ba2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ba/6ba2 https://data.pdbj.org/pub/pdb/validation_reports/ba/6ba2 ftp://data.pdbj.org/pub/pdb/validation_reports/ba/6ba2 ftp://data.pdbj.org/pub/pdb/validation_reports/ba/6ba2 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 34433.598 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: KAT8, MOF, MYST1, PP7073 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: KAT8, MOF, MYST1, PP7073 / 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌)参照: UniProt: Q9H7Z6,  ヒストンアセチルトランスフェラーゼ ヒストンアセチルトランスフェラーゼ |
|---|
-非ポリマー , 6種, 152分子 



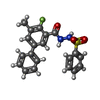






| #2: 化合物 | ChemComp-CL /  塩化物 塩化物 | ||||||
|---|---|---|---|---|---|---|---|
| #3: 化合物 | ChemComp-ZN / | ||||||
| #4: 化合物 | ChemComp-GOL /  グリセリン グリセリン#5: 化合物 | ChemComp-NA / | #6: 化合物 | ChemComp-7KM / | #7: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.31 Å3/Da / 溶媒含有率: 46.79 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7 / 詳細: 2 M NaCl, 0.1 M Tris pH 7.0, 0.22 M MgCl2 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Australian Synchrotron Australian Synchrotron  / ビームライン: MX2 / 波長: 0.9537 Å / ビームライン: MX2 / 波長: 0.9537 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2015年6月25日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9537 Å / 相対比: 1 : 0.9537 Å / 相対比: 1 |
| 反射 | 解像度: 1.85→46.23 Å / Num. obs: 27815 / % possible obs: 99.4 % / 冗長度: 4.9 % / Biso Wilson estimate: 23.5272687774 Å2 / CC1/2: 0.998 / Rmerge(I) obs: 0.072 / Rpim(I) all: 0.035 / Rrim(I) all: 0.08 / Net I/σ(I): 13.4 |
| 反射 シェル | 解像度: 1.85→1.89 Å / 冗長度: 5 % / Rmerge(I) obs: 0.698 / Mean I/σ(I) obs: 2.2 / Num. unique obs: 1669 / CC1/2: 0.741 / Rpim(I) all: 0.338 / Rrim(I) all: 0.779 / % possible all: 98.6 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: MOF Catalytic Domain 解像度: 1.85003821102→43.186116656 Å / SU ML: 0.195236071411 / 交差検証法: FREE R-VALUE / σ(F): 1.337647998 / 位相誤差: 23.9887208829
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 29.4680609733 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.85003821102→43.186116656 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj








