| 登録情報 | データベース: PDB / ID: 5yde
|
|---|
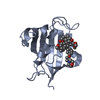
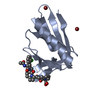
| タイトル | Crystal structure of a disease-related gene, hCDC73(1-111) |
|---|
 要素 要素 | Parafibromin |
|---|
 キーワード キーワード | TRANSCRIPTION / Tumor suppressor / cancer |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Cdc73/Paf1 complex / positive regulation of mRNA 3'-end processing / endodermal cell fate commitment / negative regulation of myeloid cell differentiation / positive regulation of cell cycle G1/S phase transition / mRNA 3'-end processing / stem cell population maintenance / negative regulation of G1/S transition of mitotic cell cycle / RNA polymerase II complex binding / positive regulation of Wnt signaling pathway ...Cdc73/Paf1 complex / positive regulation of mRNA 3'-end processing / endodermal cell fate commitment / negative regulation of myeloid cell differentiation / positive regulation of cell cycle G1/S phase transition / mRNA 3'-end processing / stem cell population maintenance / negative regulation of G1/S transition of mitotic cell cycle / RNA polymerase II complex binding / positive regulation of Wnt signaling pathway / negative regulation of fibroblast proliferation / RNA Polymerase II Transcription Elongation / Formation of RNA Pol II elongation complex / RNA Polymerase II Pre-transcription Events / transcription elongation by RNA polymerase II / regulation of cell growth / positive regulation of transcription elongation by RNA polymerase II / Hedgehog 'on' state / Formation of the beta-catenin:TCF transactivating complex / protein destabilization / Wnt signaling pathway / negative regulation of epithelial cell proliferation / E3 ubiquitin ligases ubiquitinate target proteins / cellular response to lipopolysaccharide / chromosome, telomeric region / negative regulation of cell population proliferation / negative regulation of apoptotic process / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / nucleoplasm / nucleus / cytosol類似検索 - 分子機能 Paf1 complex subunit Cdc73, N-terminal domain / Paf1 complex subunit CDC73 N-terminal / Cdc73/Parafibromin / Cell division control protein 73, C-terminal / Cell division control protein 73, C-terminal domain superfamily / RNA pol II accessory factor, Cdc73 family, C-terminal類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.023 Å 分子置換 / 解像度: 1.023 Å |
|---|
 データ登録者 データ登録者 | Sun, W. / Kuang, X.L. / Liu, Y.P. / Tian, L.F. / Yan, X.X. / Xu, W.Q. |
|---|
 引用 引用 |  ジャーナル: Sci Rep / 年: 2017 ジャーナル: Sci Rep / 年: 2017
タイトル: Crystal structure of the N-terminal domain of human CDC73 and its implications for the hyperparathyroidism-jaw tumor (HPT-JT) syndrome
著者: Sun, W. / Kuang, X.L. / Liu, Y.P. / Tian, L.F. / Yan, X.X. / Xu, W. |
|---|
| 履歴 | | 登録 | 2017年9月13日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2017年12月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年3月27日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.023 Å
分子置換 / 解像度: 1.023 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Sci Rep / 年: 2017
ジャーナル: Sci Rep / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5yde.cif.gz
5yde.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5yde.ent.gz
pdb5yde.ent.gz PDB形式
PDB形式 5yde.json.gz
5yde.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5yde_validation.pdf.gz
5yde_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5yde_full_validation.pdf.gz
5yde_full_validation.pdf.gz 5yde_validation.xml.gz
5yde_validation.xml.gz 5yde_validation.cif.gz
5yde_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/yd/5yde
https://data.pdbj.org/pub/pdb/validation_reports/yd/5yde ftp://data.pdbj.org/pub/pdb/validation_reports/yd/5yde
ftp://data.pdbj.org/pub/pdb/validation_reports/yd/5yde リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: CDC73, C1orf28, HRPT2 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CDC73, C1orf28, HRPT2 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL19U1 / 波長: 0.9785 Å
/ ビームライン: BL19U1 / 波長: 0.9785 Å 解析
解析 分子置換 / 解像度: 1.023→19.399 Å / SU ML: 0.07 / 交差検証法: FREE R-VALUE / σ(F): 1.37 / 位相誤差: 11.1
分子置換 / 解像度: 1.023→19.399 Å / SU ML: 0.07 / 交差検証法: FREE R-VALUE / σ(F): 1.37 / 位相誤差: 11.1  ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





