| 登録情報 | データベース: PDB / ID: 5t9w
|
|---|
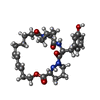
| タイトル | Discovery of a Potent Cyclophilin Inhibitor (Compound 5) based on Structural Simplification of Sanglifehrin A |
|---|
 要素 要素 | Peptidyl-prolyl cis-trans isomerase A |
|---|
 キーワード キーワード | isomerase/isomerase inhibitor / cyclophilin inhibitor antiviral HCV / isomerase-isomerase inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of protein K48-linked ubiquitination / negative regulation of viral life cycle / regulation of apoptotic signaling pathway / cell adhesion molecule production / lipid droplet organization / heparan sulfate binding / regulation of viral genome replication / virion binding / leukocyte chemotaxis / endothelial cell activation ...negative regulation of protein K48-linked ubiquitination / negative regulation of viral life cycle / regulation of apoptotic signaling pathway / cell adhesion molecule production / lipid droplet organization / heparan sulfate binding / regulation of viral genome replication / virion binding / leukocyte chemotaxis / endothelial cell activation / negative regulation of stress-activated MAPK cascade / Basigin interactions / cyclosporin A binding / Minus-strand DNA synthesis / Plus-strand DNA synthesis / Uncoating of the HIV Virion / Early Phase of HIV Life Cycle / Integration of provirus / APOBEC3G mediated resistance to HIV-1 infection / viral release from host cell / Calcineurin activates NFAT / protein peptidyl-prolyl isomerization / Binding and entry of HIV virion / positive regulation of viral genome replication / negative regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / : / neutrophil chemotaxis / activation of protein kinase B activity / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / negative regulation of protein phosphorylation / platelet aggregation / peptidylprolyl isomerase / peptidyl-prolyl cis-trans isomerase activity / positive regulation of protein secretion / negative regulation of protein kinase activity / Assembly Of The HIV Virion / Budding and maturation of HIV virion / neuron differentiation / platelet activation / SARS-CoV-1 activates/modulates innate immune responses / unfolded protein binding / integrin binding / Platelet degranulation / protein folding / positive regulation of NF-kappaB transcription factor activity / cellular response to oxidative stress / secretory granule lumen / vesicle / ficolin-1-rich granule lumen / positive regulation of MAPK cascade / positive regulation of protein phosphorylation / focal adhesion / Neutrophil degranulation / apoptotic process / protein-containing complex / RNA binding / extracellular space / extracellular exosome / extracellular region / membrane / nucleus / cytosol / cytoplasm類似検索 - 分子機能 Cyclophilin-like / Cyclophilin / Cyclophilin-type peptidyl-prolyl cis-trans isomerase / Cyclophilin-type peptidyl-prolyl cis-trans isomerase, conserved site / Cyclophilin-type peptidyl-prolyl cis-trans isomerase signature. / Cyclophilin-type peptidyl-prolyl cis-trans isomerase domain profile. / Cyclophilin-type peptidyl-prolyl cis-trans isomerase domain / Cyclophilin type peptidyl-prolyl cis-trans isomerase/CLD / Cyclophilin-like domain superfamily / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 3-[(3-hydroxyphenyl)methyl]-6-(propan-2-yl)-19-oxa-1,4,7,25-tetraazabicyclo[19.3.1]pentacosa-13,15-diene-2,5,8,20-tetrone / Chem-78E / Peptidyl-prolyl cis-trans isomerase A類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2 Å シンクロトロン / 解像度: 2 Å |
|---|
 データ登録者 データ登録者 | Appleby, T.C. / Steadman, V. / Pettit, P. / Schmitz, U. / Mackman, R.L. / Schultz, B. |
|---|
 引用 引用 |  ジャーナル: J. Med. Chem. / 年: 2017 ジャーナル: J. Med. Chem. / 年: 2017
タイトル: Discovery of Potent Cyclophilin Inhibitors Based on the Structural Simplification of Sanglifehrin A.
著者: Steadman, V.A. / Pettit, S.B. / Poullennec, K.G. / Lazarides, L. / Keats, A.J. / Dean, D.K. / Stanway, S.J. / Austin, C.A. / Sanvoisin, J.A. / Watt, G.M. / Fliri, H.G. / Liclican, A.C. / Jin, ...著者: Steadman, V.A. / Pettit, S.B. / Poullennec, K.G. / Lazarides, L. / Keats, A.J. / Dean, D.K. / Stanway, S.J. / Austin, C.A. / Sanvoisin, J.A. / Watt, G.M. / Fliri, H.G. / Liclican, A.C. / Jin, D. / Wong, M.H. / Leavitt, S.A. / Lee, Y.J. / Tian, Y. / Frey, C.R. / Appleby, T.C. / Schmitz, U. / Jansa, P. / Mackman, R.L. / Schultz, B.E. |
|---|
| 履歴 | | 登録 | 2016年9月9日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年1月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年2月8日 | Group: Database references |
|---|
| 改定 1.2 | 2017年2月15日 | Group: Database references |
|---|
| 改定 1.3 | 2017年3月29日 | Group: Structure summary |
|---|
| 改定 1.4 | 2024年3月6日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2 Å
シンクロトロン / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J. Med. Chem. / 年: 2017
ジャーナル: J. Med. Chem. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5t9w.cif.gz
5t9w.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5t9w.ent.gz
pdb5t9w.ent.gz PDB形式
PDB形式 5t9w.json.gz
5t9w.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5t9w_validation.pdf.gz
5t9w_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5t9w_full_validation.pdf.gz
5t9w_full_validation.pdf.gz 5t9w_validation.xml.gz
5t9w_validation.xml.gz 5t9w_validation.cif.gz
5t9w_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/t9/5t9w
https://data.pdbj.org/pub/pdb/validation_reports/t9/5t9w ftp://data.pdbj.org/pub/pdb/validation_reports/t9/5t9w
ftp://data.pdbj.org/pub/pdb/validation_reports/t9/5t9w リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: PPIA, CYPA / 発現宿主: Bacteria (unknown) / 参照: UniProt: P62937, peptidylprolyl isomerase
Homo sapiens (ヒト) / 遺伝子: PPIA, CYPA / 発現宿主: Bacteria (unknown) / 参照: UniProt: P62937, peptidylprolyl isomerase X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.1 / 波長: 0.979 Å
/ ビームライン: 5.0.1 / 波長: 0.979 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj