| 登録情報 | データベース: PDB / ID: 5n9m
|
|---|
| タイトル | Crystal structure of GatD - a glutamine amidotransferase from Staphylococcus aureus involved in peptidoglycan amidation |
|---|
 要素 要素 | Cobyric acid synthase |
|---|
 キーワード キーワード | TRANSFERASE / Glutamine amidotransferase Peptidoglycan amidation Multidrug resistance Staphyloccocus aureus |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
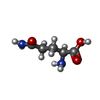
lipid II isoglutaminyl synthase (glutamine-hydrolysing) / carbon-nitrogen ligase activity on lipid II / glutaminase / cobalamin biosynthetic process / glutaminase activity / peptidoglycan biosynthetic process / cell wall organization / regulation of cell shape類似検索 - 分子機能 CobB/CobQ-like glutamine amidotransferase / Cobyric acid synthase, glutamine amidotransferase type 1 / Lipid II isoglutaminyl synthase (glutamine-hydrolyzing) subunit GatD / CobB/CobQ-like glutamine amidotransferase domain / CobBQ-type GATase domain profile. / Class I glutamine amidotransferase-like類似検索 - ドメイン・相同性 GLUTAMINE / Lipid II isoglutaminyl synthase (glutamine-hydrolyzing) subunit GatD類似検索 - 構成要素 |
|---|
| 生物種 |  Staphylococcus aureus subsp. aureus COL (黄色ブドウ球菌) Staphylococcus aureus subsp. aureus COL (黄色ブドウ球菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 1.85 Å 単波長異常分散 / 解像度: 1.85 Å |
|---|
 データ登録者 データ登録者 | Leisico, F. / Vieira, D. / Romao, M.R. / Trincao, J. / Santos-Silva, T. |
|---|
| 資金援助 |  ポルトガル, 3件 ポルトガル, 3件 | 組織 | 認可番号 | 国 |
|---|
| Fundacao para a Ciencia e Tecnologia | PD/BD/105737/2014 |  ポルトガル ポルトガル | | Fundacao para a Ciencia e Tecnologia | UID/Multi/04378/2013 |  ポルトガル ポルトガル | | Fundacao para a Ciencia e Tecnologia | PTDC/QEQ-MED/1902/2014 |  ポルトガル ポルトガル |
|
|---|
 引用 引用 |  ジャーナル: Sci Rep / 年: 2018 ジャーナル: Sci Rep / 年: 2018
タイトル: First insights of peptidoglycan amidation in Gram-positive bacteria - the high-resolution crystal structure of Staphylococcus aureus glutamine amidotransferase GatD.
著者: Leisico, F. / Vieira, D.V. / Figueiredo, T.A. / Silva, M. / Cabrita, E.J. / Sobral, R.G. / Ludovice, A.M. / Trincao, J. / Romao, M.J. / de Lencastre, H. / Santos-Silva, T. |
|---|
| 履歴 | | 登録 | 2017年2月25日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2018年3月14日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年4月11日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2019年2月20日 | Group: Advisory / Data collection / Derived calculations
カテゴリ: diffrn_source / pdbx_data_processing_status ...diffrn_source / pdbx_data_processing_status / pdbx_validate_symm_contact / struct_conn / struct_conn_type
Item: _diffrn_source.pdbx_synchrotron_site |
|---|
| 改定 1.3 | 2019年7月10日 | Group: Data collection / カテゴリ: diffrn_source / Item: _diffrn_source.pdbx_synchrotron_site |
|---|
| 改定 1.4 | 2024年5月8日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Staphylococcus aureus subsp. aureus COL (黄色ブドウ球菌)
Staphylococcus aureus subsp. aureus COL (黄色ブドウ球菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 1.85 Å
単波長異常分散 / 解像度: 1.85 Å  データ登録者
データ登録者 ポルトガル, 3件
ポルトガル, 3件  引用
引用 ジャーナル: Sci Rep / 年: 2018
ジャーナル: Sci Rep / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5n9m.cif.gz
5n9m.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5n9m.ent.gz
pdb5n9m.ent.gz PDB形式
PDB形式 5n9m.json.gz
5n9m.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5n9m_validation.pdf.gz
5n9m_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5n9m_full_validation.pdf.gz
5n9m_full_validation.pdf.gz 5n9m_validation.xml.gz
5n9m_validation.xml.gz 5n9m_validation.cif.gz
5n9m_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n9/5n9m
https://data.pdbj.org/pub/pdb/validation_reports/n9/5n9m ftp://data.pdbj.org/pub/pdb/validation_reports/n9/5n9m
ftp://data.pdbj.org/pub/pdb/validation_reports/n9/5n9m リンク
リンク 集合体
集合体


 要素
要素 Staphylococcus aureus subsp. aureus COL (黄色ブドウ球菌)
Staphylococcus aureus subsp. aureus COL (黄色ブドウ球菌)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I02 / 波長: 1 Å
/ ビームライン: I02 / 波長: 1 Å 解析
解析 単波長異常分散 / 解像度: 1.85→36.435 Å / SU ML: 0.21 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 17.59
単波長異常分散 / 解像度: 1.85→36.435 Å / SU ML: 0.21 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 17.59  ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj