+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5med | ||||||
|---|---|---|---|---|---|---|---|
| Title | Cyanothece lipoxygenase 2 (CspLOX2) | ||||||
 Components Components | Arachidonate 15-lipoxygenase | ||||||
 Keywords Keywords | OXIDOREDUCTASE / Linoleate 11-lipoxygenase | ||||||
| Function / homology |  Function and homology information Function and homology informationarachidonate 15-lipoxygenase / arachidonate 15-lipoxygenase activity / lipid oxidation / metal ion binding Similarity search - Function | ||||||
| Biological species | Cyanothece sp. | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.8 Å MOLECULAR REPLACEMENT / Resolution: 1.8 Å | ||||||
 Authors Authors | Newie, J. / Neumann, P. / Werner, M. / Mata, R.A. / Ficner, R. / Feussner, I. | ||||||
 Citation Citation |  Journal: Sci Rep / Year: 2017 Journal: Sci Rep / Year: 2017Title: Lipoxygenase 2 from Cyanothece sp. controls dioxygen insertion by steric shielding and substrate fixation. Authors: Newie, J. / Neumann, P. / Werner, M. / Mata, R.A. / Ficner, R. / Feussner, I. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5med.cif.gz 5med.cif.gz | 266 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5med.ent.gz pdb5med.ent.gz | 210.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5med.json.gz 5med.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/me/5med https://data.pdbj.org/pub/pdb/validation_reports/me/5med ftp://data.pdbj.org/pub/pdb/validation_reports/me/5med ftp://data.pdbj.org/pub/pdb/validation_reports/me/5med | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5meeC  5mefC  5megC  3o8yS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 64557.562 Da / Num. of mol.: 2 / Fragment: residues 1-569 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Cyanothece sp. (strain PCC 8801) (bacteria) Cyanothece sp. (strain PCC 8801) (bacteria)Gene: PCC8801_3106 / Plasmid: pET28a / Details (production host): N-terminal His6-tag / Production host:  |
|---|
-Non-polymers , 9 types, 1065 molecules 
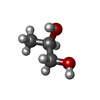
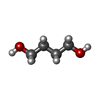


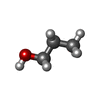
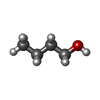









| #2: Chemical | | #3: Chemical | ChemComp-PGR / #4: Chemical | ChemComp-BU1 / #5: Chemical | ChemComp-GOL / | #6: Chemical | ChemComp-HEZ / | #7: Chemical | #8: Chemical | ChemComp-1BO / | #9: Chemical | #10: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.9 Å3/Da / Density % sol: 57.6 % |
|---|---|
| Crystal grow | Temperature: 277 K / Method: vapor diffusion, sitting drop / pH: 6.5 Details: 10% PEG 4000, 20% glycerol, 0.1 M MES/imidazole, 0.02 M of each alcohol (1,6-hexanediol, 1-butanol, 1,2-propanediol, 2-propanol, 1,4-butanediol, 1,3-propanediol) |
-Data collection
| Diffraction | Mean temperature: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  BESSY BESSY  / Beamline: 14.1 / Wavelength: 0.9184 Å / Beamline: 14.1 / Wavelength: 0.9184 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: MARMOSAIC 225 mm CCD / Detector: CCD / Date: Nov 22, 2012 / Details: mirrors | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Monochromator: GRAPHITE, / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9184 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.8→45 Å / Num. obs: 134076 / % possible obs: 95.7 % / Observed criterion σ(I): -3 / Redundancy: 3.2 % / Biso Wilson estimate: 24.48 Å2 / CC1/2: 0.996 / Rmerge(I) obs: 0.071 / Net I/σ(I): 10.1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell |
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3o8y Resolution: 1.8→41.553 Å / SU ML: 0.19 / Cross valid method: FREE R-VALUE / σ(F): 1.36 / Phase error: 20.23
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 1.1 Å / VDW probe radii: 1.3 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 114.29 Å2 / Biso mean: 30.3083 Å2 / Biso min: 11.58 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.8→41.553 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 30
|
 Movie
Movie Controller
Controller













 PDBj
PDBj










