| 登録情報 | データベース: PDB / ID: 5bqi
|
|---|
| タイトル | Discovery of a Potent and Selective mPGES-1 Inhibitor for the Treatment of Pain |
|---|
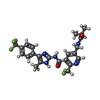
 要素 要素 | Prostaglandin E synthase |
|---|
 キーワード キーワード | Isomerase/isomerase inhibitor / mPGES-1 / enzyme / integral membrane protein / TARGETNAME / Isomerase-isomerase inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
prostaglandin-E synthase / prostaglandin-E synthase activity / regulation of fever generation / prostaglandin-D synthase activity / glutathione binding / positive regulation of prostaglandin secretion / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / glutathione peroxidase activity / prostaglandin biosynthetic process / prostaglandin metabolic process ...prostaglandin-E synthase / prostaglandin-E synthase activity / regulation of fever generation / prostaglandin-D synthase activity / glutathione binding / positive regulation of prostaglandin secretion / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / glutathione peroxidase activity / prostaglandin biosynthetic process / prostaglandin metabolic process / nuclear envelope lumen / glutathione transferase / glutathione transferase activity / 酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / sensory perception of pain / regulation of inflammatory response / cell population proliferation / negative regulation of cell population proliferation / endoplasmic reticulum membrane / perinuclear region of cytoplasm / signal transduction / membrane類似検索 - 分子機能 Microsomal glutathione S-transferase 1-like / Membrane associated eicosanoid/glutathione metabolism-like domain / Membrane-associated, eicosanoid/glutathione metabolism (MAPEG) protein / Membrane associated eicosanoid/glutathione metabolism-like domain superfamily / MAPEG family / Four Helix Bundle (Hemerythrin (Met), subunit A) / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 (2-hydroxyethoxy)acetaldehyde / Chem-4UL / GLUTATHIONE / Prostaglandin E synthase類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.88 Å シンクロトロン / 解像度: 1.88 Å |
|---|
 データ登録者 データ登録者 | Fisher, M.J. / Schiffler, M.A. / Kuklish, S.L. / Antonysamy, S. / Luz, J.G. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2016 ジャーナル: J.Med.Chem. / 年: 2016
タイトル: Discovery and Characterization of 2-Acylaminoimidazole Microsomal Prostaglandin E Synthase-1 Inhibitors.
著者: Schiffler, M.A. / Antonysamy, S. / Bhattachar, S.N. / Campanale, K.M. / Chandrasekhar, S. / Condon, B. / Desai, P.V. / Fisher, M.J. / Groshong, C. / Harvey, A. / Hickey, M.J. / Hughes, N.E. / ...著者: Schiffler, M.A. / Antonysamy, S. / Bhattachar, S.N. / Campanale, K.M. / Chandrasekhar, S. / Condon, B. / Desai, P.V. / Fisher, M.J. / Groshong, C. / Harvey, A. / Hickey, M.J. / Hughes, N.E. / Jones, S.A. / Kim, E.J. / Kuklish, S.L. / Luz, J.G. / Norman, B.H. / Rathmell, R.E. / Rizzo, J.R. / Seng, T.W. / Thibodeaux, S.J. / Woods, T.A. / York, J.S. / Yu, X.P. |
|---|
| 履歴 | | 登録 | 2015年5月29日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年4月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年7月29日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp / citation ...chem_comp / citation / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / pdbx_struct_oper_list / struct_site / struct_site_gen
Item: _chem_comp.mon_nstd_flag / _chem_comp.name ..._chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _citation.journal_id_CSD / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _pdbx_struct_oper_list.symmetry_operation
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 1.2 | 2024年3月6日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 1.88 Å
シンクロトロン / 解像度: 1.88 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2016
ジャーナル: J.Med.Chem. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5bqi.cif.gz
5bqi.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5bqi.ent.gz
pdb5bqi.ent.gz PDB形式
PDB形式 5bqi.json.gz
5bqi.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5bqi_validation.pdf.gz
5bqi_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5bqi_full_validation.pdf.gz
5bqi_full_validation.pdf.gz 5bqi_validation.xml.gz
5bqi_validation.xml.gz 5bqi_validation.cif.gz
5bqi_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/bq/5bqi
https://data.pdbj.org/pub/pdb/validation_reports/bq/5bqi ftp://data.pdbj.org/pub/pdb/validation_reports/bq/5bqi
ftp://data.pdbj.org/pub/pdb/validation_reports/bq/5bqi リンク
リンク 集合体
集合体

 要素
要素

 Homo sapiens (ヒト) / 遺伝子: PTGES, MGST1L1, MPGES1, PGES, PIG12
Homo sapiens (ヒト) / 遺伝子: PTGES, MGST1L1, MPGES1, PGES, PIG12







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 31-ID / 波長: 0.97931 Å
/ ビームライン: 31-ID / 波長: 0.97931 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj






