[English] 日本語
 Yorodumi
Yorodumi- PDB-4wy2: Crystal structure of universal stress protein E from Proteus mira... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4wy2 | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of universal stress protein E from Proteus mirabilis in complex with UDP-3-O-[(3R)-3-hydroxytetradecanoyl]-N-acetyl-alpha-glucosamine | ||||||
 Components Components | Universal stress protein E | ||||||
 Keywords Keywords | STRUCTURAL GENOMICS / UNKNOWN FUNCTION / MCSG / PSI-Biology / Midwest Center for Structural Genomics | ||||||
| Function / homology | Rossmann fold - #12370 / UspA / Universal stress protein family / Rossmann fold / 3-Layer(aba) Sandwich / cytoplasm / Alpha Beta / Chem-U20 / Universal stress protein E Function and homology information Function and homology information | ||||||
| Biological species |  Proteus mirabilis (bacteria) Proteus mirabilis (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.8 Å MOLECULAR REPLACEMENT / Resolution: 1.8 Å | ||||||
 Authors Authors | Shumilin, I.A. / Shabalin, I.G. / Handing, K.B. / Joachimiak, A. / Minor, W. / Midwest Center for Structural Genomics (MCSG) | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: to be published Journal: to be publishedTitle: Crystal structure of universal stress protein E from Proteus mirabilis incomplex withUDP-3-O-[(3R)-3-hydroxytetradecanoyl]-N-acetyl-alpha-glucosamine Authors: Shumilin, I.A. / Minor, W. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4wy2.cif.gz 4wy2.cif.gz | 152.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4wy2.ent.gz pdb4wy2.ent.gz | 116.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4wy2.json.gz 4wy2.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/wy/4wy2 https://data.pdbj.org/pub/pdb/validation_reports/wy/4wy2 ftp://data.pdbj.org/pub/pdb/validation_reports/wy/4wy2 ftp://data.pdbj.org/pub/pdb/validation_reports/wy/4wy2 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  3olqS S: Starting model for refinement |
|---|---|
| Similar structure data | |
| Other databases |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
| ||||||||
| Details | biological unit is the same as asym. |
- Components
Components
-Protein , 1 types, 1 molecules A
| #1: Protein | Mass: 36269.566 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Proteus mirabilis (bacteria) / Strain: HI4320 / Gene: uspE, PMI1202 / Plasmid: pMCSG7 / Production host: Proteus mirabilis (bacteria) / Strain: HI4320 / Gene: uspE, PMI1202 / Plasmid: pMCSG7 / Production host:  |
|---|
-Non-polymers , 5 types, 323 molecules 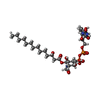








| #2: Chemical | ChemComp-U20 / | ||||||
|---|---|---|---|---|---|---|---|
| #3: Chemical | | #4: Chemical | ChemComp-CL / | #5: Chemical | ChemComp-SO4 / | #6: Water | ChemComp-HOH / | |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3 Å3/Da / Density % sol: 59.07 % |
|---|---|
| Crystal grow | Temperature: 289 K / Method: vapor diffusion / pH: 6.5 Details: 0.2 ul of 12 mg/ml protein in 10mM HEPES, 500mM NaCl pH 7.5 were mixed with 0.2 ul of the Quiagen Cryos Suite condition #50 (1.36 M Ammonium Sulfate, 0.085 M MES pH 6.5, 8.5% Dioxane, 15% ...Details: 0.2 ul of 12 mg/ml protein in 10mM HEPES, 500mM NaCl pH 7.5 were mixed with 0.2 ul of the Quiagen Cryos Suite condition #50 (1.36 M Ammonium Sulfate, 0.085 M MES pH 6.5, 8.5% Dioxane, 15% Glycerol) and equilibrated against the mother liquor in 96 Well 2 drop Crystallization Plate (MRC) PH range: 6.5-7.5 |
-Data collection
| Diffraction | Mean temperature: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 21-ID-F / Wavelength: 0.97872 Å / Beamline: 21-ID-F / Wavelength: 0.97872 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: MARMOSAIC 225 mm CCD / Detector: CCD / Date: Jul 24, 2014 / Details: Beryllium Lenses | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Monochromator: Diamond [111] / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.97872 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.8→50 Å / Num. obs: 41254 / % possible obs: 99.6 % / Observed criterion σ(I): -3 / Redundancy: 6.6 % / Biso Wilson estimate: 29 Å2 / Rmerge(I) obs: 0.08 / Rpim(I) all: 0.033 / Rrim(I) all: 0.087 / Χ2: 1.281 / Net I/av σ(I): 22.433 / Net I/σ(I): 9.5 / Num. measured all: 274114 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1 / Rejects: _
|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3OLQ Resolution: 1.8→50 Å / Cor.coef. Fo:Fc: 0.969 / Cor.coef. Fo:Fc free: 0.955 / WRfactor Rfree: 0.2066 / WRfactor Rwork: 0.1721 / FOM work R set: 0.8453 / SU B: 4.761 / SU ML: 0.077 / SU R Cruickshank DPI: 0.1016 / SU Rfree: 0.1003 / Cross valid method: THROUGHOUT / σ(F): 0 / ESU R: 0.102 / ESU R Free: 0.1 / Stereochemistry target values: MAXIMUM LIKELIHOOD Details: U VALUES : WITH TLS ADDED HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 189.93 Å2 / Biso mean: 35.365 Å2 / Biso min: 13.96 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.8→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.8→1.847 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller










 PDBj
PDBj



