| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| MxCuBE | | データ収集 | | PHASER | | 位相決定 | | PHENIX | (phenix.refine: 1.8.2_1309)| 精密化 | | XDS | | データ削減 | | XSCALE | | データスケーリング | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: PDB ENTRY 3RLB
解像度: 2.4→42.304 Å / SU ML: 0.39 / σ(F): 1.34 / 位相誤差: 28.2 / 立体化学のターゲット値: ML
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.2568 | 1296 | 5 % |
|---|
| Rwork | 0.2044 | - | - |
|---|
| obs | 0.2071 | 25926 | 99.26 % |
|---|
| all | - | 25938 | - |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL |
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 2.4→42.304 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 2732 | 0 | 360 | 46 | 3138 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.014 | 3171 | | X-RAY DIFFRACTION | f_angle_d| 1.48 | 4208 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 19.972 | 1276 | | X-RAY DIFFRACTION | f_chiral_restr| 0.077 | 466 | | X-RAY DIFFRACTION | f_plane_restr| 0.008 | 452 | | | | | |
|
|---|
| LS精密化 シェル | | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Refine-ID | % reflection obs (%) |
|---|
| 2.4-2.4961 | 0.3564 | 142 | 0.289 | 2704 | X-RAY DIFFRACTION | 99 | | 2.4961-2.6097 | 0.3424 | 144 | 0.2708 | 2728 | X-RAY DIFFRACTION | 100 | | 2.6097-2.7473 | 0.3809 | 144 | 0.2421 | 2738 | X-RAY DIFFRACTION | 100 | | 2.7473-2.9194 | 0.2942 | 144 | 0.2183 | 2739 | X-RAY DIFFRACTION | 99 | | 2.9194-3.1447 | 0.2349 | 143 | 0.2102 | 2707 | X-RAY DIFFRACTION | 99 | | 3.1447-3.461 | 0.1961 | 144 | 0.1711 | 2748 | X-RAY DIFFRACTION | 100 | | 3.461-3.9615 | 0.2853 | 143 | 0.1822 | 2720 | X-RAY DIFFRACTION | 99 | | 3.9615-4.9899 | 0.2382 | 145 | 0.1725 | 2749 | X-RAY DIFFRACTION | 99 | | 4.9899-42.3108 | 0.2247 | 147 | 0.2266 | 2797 | X-RAY DIFFRACTION | 99 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 7.7869 | 1.9262 | -0.8354 | 5.2324 | -6.5282 | 9.553 | -0.4479 | -1.209 | -0.6226 | 0.8506 | -0.9204 | -1.6362 | 0.348 | 0.9923 | 1.2842 | 0.5489 | 0.0401 | 0.1706 | 0.6838 | 0.0637 | 0.438 | 32.7223 | 3.0002 | 55.3128 | | 2 | 9.1453 | -4.6166 | 4.1951 | 9.8248 | -6.9597 | 5.109 | -0.0782 | -1.1532 | 0.3132 | 1.7656 | 0.573 | 0.0476 | -1.5392 | -0.5618 | -0.3822 | 0.8537 | 0.0152 | 0.033 | 0.4083 | -0.0734 | 0.5822 | 27.541 | 21.3992 | 43.1215 | | 3 | 2.6921 | 1.0357 | -0.1199 | 6.8761 | 0.1718 | 4.3082 | -0.2479 | -0.3667 | -0.2378 | 1.1468 | 0.1583 | -0.1749 | -0.0325 | 0.0419 | 0.0652 | 0.3865 | 0.0737 | -0.0772 | 0.4115 | 0.0514 | 0.3383 | 26.6685 | 3.6816 | 50.9388 | | 4 | 8.1495 | -3.838 | -3.3753 | 8.2222 | 3.6105 | 6.8091 | -0.0621 | -0.4039 | 0.4959 | 0.5295 | 0.0046 | -0.2185 | -0.4611 | -0.2214 | 0.1238 | 0.4638 | 0.0139 | 0.0225 | 0.3162 | 0.0632 | 0.3677 | 18.5737 | 10.6222 | 50.6132 | | 5 | 5.9969 | -3.8725 | 0.9033 | 3.8748 | -4.1426 | 9.1723 | 0.3053 | 0.0389 | -1.0344 | 0.2105 | -0.4872 | 1.3756 | 0.4991 | 0.3137 | 0.2777 | 0.8096 | 0.0586 | 0.0367 | 0.3891 | 0.0262 | 0.7294 | 33.0062 | -9.53 | 38.1109 | | 6 | 6.7423 | -5.9789 | -0.9058 | 9.7373 | 0.9076 | 2.7456 | 0.1819 | -0.2693 | -0.4842 | -0.2116 | -0.1855 | 0.6942 | -0.1065 | -0.3441 | 0.0012 | 0.3006 | -0.0467 | 0.0133 | 0.3133 | 0.0194 | 0.3868 | 18.7928 | 8.9257 | 40.0775 | | 7 | 6.2667 | -3.6074 | -1.3677 | 6.5122 | -1.4251 | 4.0481 | 0.2329 | 0.193 | 0.5903 | -0.1183 | -0.0201 | -0.1396 | -0.6438 | -0.1543 | -0.1996 | 0.4585 | -0.0359 | -0.061 | 0.2652 | -0.0325 | 0.3985 | 23.6126 | 16.5352 | 37.8784 | | 8 | 9.6212 | -1.4631 | -6.3705 | 0.2667 | 1.0284 | 4.3032 | -0.4742 | -1.0751 | -1.7293 | 1.5823 | 0.2112 | -1.5295 | 1.5182 | 1.4075 | 0.2783 | 0.856 | 0.1931 | -0.2846 | 0.5061 | 0.0274 | 0.7561 | 39.6116 | -3.795 | 42.3195 | | 9 | 2.8391 | 2.2674 | 1.3592 | 9.4845 | -1.2633 | 1.3643 | -0.7478 | -0.7408 | -0.9871 | 1.8786 | -0.2133 | -1.8485 | 1.9564 | 0.808 | -0.1365 | 1.8749 | 0.9401 | 0.1597 | 0.141 | 0.2936 | 0.9579 | 37.5104 | -13.3824 | 49.1755 | | 10 | 5.7031 | -1.3212 | 2.0026 | 5.4016 | -4.6592 | 4.6832 | -0.6473 | 1.0822 | 0.2016 | -1.9353 | -0.4689 | -1.2888 | 0.2444 | 0.4345 | 1.0634 | 0.5816 | -0.0251 | 0.0833 | 0.7742 | 0.0439 | 0.5725 | 37.2155 | 19.8073 | 7.6101 | | 11 | 4.3066 | 3.9367 | 1.5408 | 9.6248 | -1.8696 | 2.8993 | -0.4299 | 0.7719 | -0.7404 | -1.3278 | 0.0146 | -0.7178 | 1.0915 | 0.3775 | 0.3375 | 0.7358 | 0.0007 | 0.1751 | 0.376 | -0.1204 | 0.5839 | 31.466 | 1.5304 | 20.521 | | 12 | 0.9198 | -1.3591 | -0.445 | 3.8786 | -0.7528 | 4.9525 | -0.3695 | 0.2296 | -0.0911 | -0.4769 | 0.2337 | -0.6339 | -0.004 | 0.0064 | 0.1097 | 0.3264 | -0.1426 | 0.0451 | 0.4746 | 0.0349 | 0.4479 | 31.1001 | 19.162 | 11.9807 | | 13 | 8.2557 | 4.431 | 2.3608 | 3.7658 | 3.4091 | 6.934 | -0.0251 | 0.4029 | -0.7916 | -0.8379 | -0.1078 | -1.1282 | 0.3945 | -0.4186 | 0.1197 | 0.5987 | -0.071 | 0.0064 | 0.3454 | 0.0162 | 0.4136 | 22.848 | 12.3809 | 12.4235 | | 14 | 5.2967 | 5.9505 | 7.0231 | 6.6988 | 7.8593 | 9.3148 | -0.3996 | 0.2109 | 0.5294 | 0.1552 | 0.1188 | 1.2434 | -0.6156 | 0.4261 | 0.3474 | 0.8214 | -0.1218 | -0.065 | 0.4553 | 0.025 | 0.6902 | 37.142 | 33.8482 | 23.7656 | | 15 | 4.6586 | 4.7889 | 3.9091 | 9.0478 | 1.3195 | 5.1569 | 0.0363 | -0.1708 | -0.0516 | -0.1865 | -0.1185 | -0.4009 | -0.3927 | -0.151 | 0.0371 | 0.279 | 0.0012 | 0.023 | 0.3681 | -0.0021 | 0.4433 | 23.4579 | 14.9605 | 23.0808 | | 16 | 7.0359 | 5.0448 | -4.3636 | 4.9866 | -5.2109 | 5.8461 | -0.4886 | -0.04 | -0.4384 | -0.4847 | -0.0964 | -0.3873 | 0.5501 | -0.5052 | 0.5609 | 0.7703 | -0.1212 | -0.0076 | 0.3199 | -0.0077 | 0.7667 | 17.2184 | -4.2099 | 23.5587 | | 17 | 3.6095 | 1.9607 | -1.0891 | 5.0588 | -2.698 | 5.402 | -0.1129 | 0.0457 | -0.3169 | 0.0973 | -0.3942 | -1.1231 | -0.3143 | 0.6703 | 0.4829 | 0.3856 | 0.0039 | -0.0594 | 0.3478 | 0.0391 | 0.5558 | 33.9111 | 14.0687 | 25.3608 | | 18 | 6.5159 | 4.9036 | -0.4514 | 5.9727 | -1.0829 | 0.2696 | -1.577 | 1.419 | 0.6759 | -2.1838 | -0.1033 | 0.2161 | -1.4962 | 0.7617 | 0.6657 | 1.7048 | -0.7199 | -0.3892 | 0.6012 | 0.1203 | 1.1429 | 42.1711 | 36.2735 | 13.5901 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details |
|---|
| 1 | X-RAY DIFFRACTION | 1 | chain 'A' and (resid 7 through 26 )| 2 | X-RAY DIFFRACTION | 2 | chain 'A' and (resid 27 through 38 )| 3 | X-RAY DIFFRACTION | 3 | chain 'A' and (resid 39 through 69 )| 4 | X-RAY DIFFRACTION | 4 | chain 'A' and (resid 70 through 98 )| 5 | X-RAY DIFFRACTION | 5 | chain 'A' and (resid 99 through 107 )| 6 | X-RAY DIFFRACTION | 6 | chain 'A' and (resid 108 through 132 )| 7 | X-RAY DIFFRACTION | 7 | chain 'A' and (resid 133 through 172 )| 8 | X-RAY DIFFRACTION | 8 | chain 'A' and (resid 173 through 177 )| 9 | X-RAY DIFFRACTION | 9 | chain 'A' and (resid 178 through 182 )| 10 | X-RAY DIFFRACTION | 10 | chain 'B' and (resid 7 through 26 )| 11 | X-RAY DIFFRACTION | 11 | chain 'B' and (resid 27 through 38 )| 12 | X-RAY DIFFRACTION | 12 | chain 'B' and (resid 39 through 69 )| 13 | X-RAY DIFFRACTION | 13 | chain 'B' and (resid 70 through 98 )| 14 | X-RAY DIFFRACTION | 14 | chain 'B' and (resid 99 through 106 )| 15 | X-RAY DIFFRACTION | 15 | chain 'B' and (resid 107 through 132 )| 16 | X-RAY DIFFRACTION | 16 | chain 'B | | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Lactococcus lactis (乳酸菌)
Lactococcus lactis (乳酸菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.4 Å
分子置換 / 解像度: 2.4 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4n4d.cif.gz
4n4d.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4n4d.ent.gz
pdb4n4d.ent.gz PDB形式
PDB形式 4n4d.json.gz
4n4d.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4n4d_validation.pdf.gz
4n4d_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4n4d_full_validation.pdf.gz
4n4d_full_validation.pdf.gz 4n4d_validation.xml.gz
4n4d_validation.xml.gz 4n4d_validation.cif.gz
4n4d_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d
https://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d ftp://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d
ftp://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d
 リンク
リンク 集合体
集合体

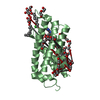
 要素
要素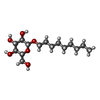

 Lactococcus lactis (乳酸菌) / 株: NZ9000 / 遺伝子: LLNZ_01755 / 発現宿主:
Lactococcus lactis (乳酸菌) / 株: NZ9000 / 遺伝子: LLNZ_01755 / 発現宿主:  Lactococcus lactis (乳酸菌) / 株 (発現宿主): NZ9000 / 参照: UniProt: D8KFM5
Lactococcus lactis (乳酸菌) / 株 (発現宿主): NZ9000 / 参照: UniProt: D8KFM5















 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID23-1 / 波長: 0.97625 Å
/ ビームライン: ID23-1 / 波長: 0.97625 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







