| Entry | Database: PDB / ID: 4n4d
|
|---|
| Title | Structure of ThiT with AV-38 bound |
|---|
 Components Components | thiamine binding protein ThiT |
|---|
 Keywords Keywords | THIAMINE BINDING PROTEIN/INHIBITOR / S-component / ECF transporter / ABC transporter / transport protein / THIAMINE BINDING PROTEIN-INHIBITOR complex |
|---|
| Function / homology | Arp2/3 complex 21 kDa subunit ARPC3 - #20 / Arp2/3 complex 21 kDa subunit ARPC3 / Orthogonal Bundle / Mainly Alpha / DI(HYDROXYETHYL)ETHER / TRIETHYLENE GLYCOL / Chem-XX8 / :  Function and homology information Function and homology information |
|---|
| Biological species |  Lactococcus lactis (lactic acid bacteria) Lactococcus lactis (lactic acid bacteria) |
|---|
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.4 Å MOLECULAR REPLACEMENT / Resolution: 2.4 Å |
|---|
 Authors Authors | Swier, L.J.Y.M. / Guskov, A. / Slotboom, D.J. |
|---|
 Citation Citation |  Journal: To be Published Journal: To be Published
Title: Structural studies on the thiamine binding protein ThiT
Authors: Swier, L.J.Y.M. / Gomez, L. / Guskov, A. / Hirsch, A.K.H. / Slotboom, D.J. |
|---|
| History | | Deposition | Oct 8, 2013 | Deposition site: RCSB / Processing site: RCSB |
|---|
| Revision 1.0 | Sep 17, 2014 | Provider: repository / Type: Initial release |
|---|
| Revision 1.1 | Jul 29, 2020 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
Category: chem_comp / entity ...chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_ref_seq_dif / struct_site / struct_site_gen
Item: _chem_comp.mon_nstd_flag / _chem_comp.name ..._chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.pdbx_synonyms / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_ref_seq_dif.details
Description: Carbohydrate remediation / Provider: repository / Type: Remediation |
|---|
| Revision 1.2 | Sep 20, 2023 | Group: Data collection / Database references ...Data collection / Database references / Refinement description / Structure summary
Category: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information Lactococcus lactis (lactic acid bacteria)
Lactococcus lactis (lactic acid bacteria) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.4 Å
MOLECULAR REPLACEMENT / Resolution: 2.4 Å  Authors
Authors Citation
Citation Journal: To be Published
Journal: To be Published Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 4n4d.cif.gz
4n4d.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb4n4d.ent.gz
pdb4n4d.ent.gz PDB format
PDB format 4n4d.json.gz
4n4d.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d
https://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d ftp://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d
ftp://data.pdbj.org/pub/pdb/validation_reports/n4/4n4d
 Links
Links Assembly
Assembly

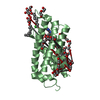
 Components
Components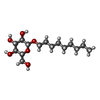

 Lactococcus lactis (lactic acid bacteria)
Lactococcus lactis (lactic acid bacteria) Lactococcus lactis (lactic acid bacteria) / Strain (production host): NZ9000 / References: UniProt: D8KFM5
Lactococcus lactis (lactic acid bacteria) / Strain (production host): NZ9000 / References: UniProt: D8KFM5















 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  ESRF
ESRF  / Beamline: ID23-1 / Wavelength: 0.97625 Å
/ Beamline: ID23-1 / Wavelength: 0.97625 Å Processing
Processing MOLECULAR REPLACEMENT
MOLECULAR REPLACEMENT Movie
Movie Controller
Controller













 PDBj
PDBj







