[English] 日本語
 Yorodumi
Yorodumi- PDB-4lv6: Crystal Structure of small molecule disulfide 4 covalently bound ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4lv6 | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of small molecule disulfide 4 covalently bound to K-Ras G12C | ||||||
 Components Components | GTPase KRas | ||||||
 Keywords Keywords | SIGNALING PROTEIN/INHIBITOR / GTPase / GDP bound / small molecule inhibitor / covalent binder / SIGNALING PROTEIN-INHIBITOR complex | ||||||
| Function / homology |  Function and homology information Function and homology informationresponse to mineralocorticoid / GMP binding / forebrain astrocyte development / LRR domain binding / regulation of synaptic transmission, GABAergic / negative regulation of epithelial cell differentiation / response to isolation stress / response to gravity / epithelial tube branching involved in lung morphogenesis / type I pneumocyte differentiation ...response to mineralocorticoid / GMP binding / forebrain astrocyte development / LRR domain binding / regulation of synaptic transmission, GABAergic / negative regulation of epithelial cell differentiation / response to isolation stress / response to gravity / epithelial tube branching involved in lung morphogenesis / type I pneumocyte differentiation / Rac protein signal transduction / Signaling by RAS GAP mutants / Signaling by RAS GTPase mutants / positive regulation of Rac protein signal transduction / Activation of RAS in B cells / myoblast proliferation / RAS signaling downstream of NF1 loss-of-function variants / RUNX3 regulates p14-ARF / positive regulation of glial cell proliferation / skeletal muscle cell differentiation / SOS-mediated signalling / Activated NTRK3 signals through RAS / Activated NTRK2 signals through RAS / SHC1 events in ERBB4 signaling / cardiac muscle cell proliferation / Signalling to RAS / SHC-related events triggered by IGF1R / Activated NTRK2 signals through FRS2 and FRS3 / Estrogen-stimulated signaling through PRKCZ / SHC-mediated cascade:FGFR3 / glial cell proliferation / MET activates RAS signaling / SHC-mediated cascade:FGFR2 / SHC-mediated cascade:FGFR4 / PTK6 Regulates RHO GTPases, RAS GTPase and MAP kinases / Signaling by PDGFRA transmembrane, juxtamembrane and kinase domain mutants / Signaling by PDGFRA extracellular domain mutants / Erythropoietin activates RAS / SHC-mediated cascade:FGFR1 / Signaling by FGFR4 in disease / Signaling by CSF3 (G-CSF) / FRS-mediated FGFR3 signaling / Signaling by FLT3 ITD and TKD mutants / FRS-mediated FGFR2 signaling / FRS-mediated FGFR4 signaling / p38MAPK events / FRS-mediated FGFR1 signaling / Signaling by FGFR3 in disease / protein-membrane adaptor activity / striated muscle cell differentiation / Tie2 Signaling / Signaling by FGFR2 in disease / GRB2 events in EGFR signaling / Signaling by FLT3 fusion proteins / SHC1 events in EGFR signaling / FLT3 Signaling / Signaling by FGFR1 in disease / EGFR Transactivation by Gastrin / NCAM signaling for neurite out-growth / CD209 (DC-SIGN) signaling / GRB2 events in ERBB2 signaling / homeostasis of number of cells within a tissue / Downstream signal transduction / Insulin receptor signalling cascade / SHC1 events in ERBB2 signaling / Ras activation upon Ca2+ influx through NMDA receptor / response to glucocorticoid / Constitutive Signaling by Overexpressed ERBB2 / Signaling by phosphorylated juxtamembrane, extracellular and kinase domain KIT mutants / VEGFR2 mediated cell proliferation / small monomeric GTPase / FCERI mediated MAPK activation / liver development / Signaling by ERBB2 TMD/JMD mutants / female pregnancy / RAF activation / Signaling by SCF-KIT / Constitutive Signaling by EGFRvIII / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / Signaling by ERBB2 ECD mutants / Signaling by ERBB2 KD Mutants / regulation of long-term neuronal synaptic plasticity / visual learning / cytoplasmic side of plasma membrane / cytokine-mediated signaling pathway / Signaling by CSF1 (M-CSF) in myeloid cells / Regulation of RAS by GAPs / Negative regulation of MAPK pathway / Signaling by RAF1 mutants / RAS processing / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / GDP binding / Signaling by BRAF and RAF1 fusions / positive regulation of cellular senescence / DAP12 signaling / MAPK cascade / Constitutive Signaling by Ligand-Responsive EGFR Cancer Variants Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 1.5 Å molecular replacement / Resolution: 1.5 Å | ||||||
 Authors Authors | Ostrem, J.M. / Peters, U. / Sos, M.L. / Wells, J.A. / Shokat, K.M. | ||||||
 Citation Citation |  Journal: Nature / Year: 2013 Journal: Nature / Year: 2013Title: K-Ras(G12C) inhibitors allosterically control GTP affinity and effector interactions. Authors: Ostrem, J.M. / Peters, U. / Sos, M.L. / Wells, J.A. / Shokat, K.M. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4lv6.cif.gz 4lv6.cif.gz | 219.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4lv6.ent.gz pdb4lv6.ent.gz | 178.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4lv6.json.gz 4lv6.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/lv/4lv6 https://data.pdbj.org/pub/pdb/validation_reports/lv/4lv6 ftp://data.pdbj.org/pub/pdb/validation_reports/lv/4lv6 ftp://data.pdbj.org/pub/pdb/validation_reports/lv/4lv6 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4l8gC  4l9sC  4l9wC  4lpkC  4lrwC  4lucC  4lyfC  4lyhC  4lyjC  4m1oC  4m1sC  4m1tC  4m1wC  4m1yC  4m21C  4m22C C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 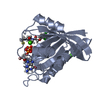
| ||||||||
| 3 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 19352.785 Da / Num. of mol.: 2 / Fragment: UNP residues 1-169 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: KRAS, KRAS isoform 2B, KRAS2, RASK2 / Plasmid: pJexpress411 / Production host: Homo sapiens (human) / Gene: KRAS, KRAS isoform 2B, KRAS2, RASK2 / Plasmid: pJexpress411 / Production host:  #2: Chemical | ChemComp-CA / #3: Chemical | #4: Chemical | #5: Water | ChemComp-HOH / | Has protein modification | Y | Sequence details | THE SEQUENCE IN THE STRUCTRUE REPRESENTS ISOFORM 2B OF GTPASE KRAS. THIS ISOFORM DIFFERS FROM THE ...THE SEQUENCE IN THE STRUCTRUE REPRESENTS | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2 Å3/Da / Density % sol: 38.5 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 7.5 Details: 22% PEG8000, 0.1M CaCl2, 0.1M TRIS, pH 7.5, vapor diffusion, hanging drop, temperature 293K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ALS ALS  / Beamline: 8.2.1 / Wavelength: 1 Å / Beamline: 8.2.1 / Wavelength: 1 Å |
| Detector | Type: ADSC QUANTUM 315r / Detector: CCD / Date: Dec 17, 2010 |
| Diffraction measurement | Details: 1.00 degrees, 3.0 sec, detector distance 175.00 mm / Method: \w scans |
| Radiation | Monochromator: Double crystal, Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Av R equivalents: 0.068 / Number: 179388 |
| Reflection | Resolution: 1.5→20 Å / Num. obs: 46311 / % possible obs: 96.3 % / Observed criterion σ(F): 0 / Observed criterion σ(I): -3 / Redundancy: 3.9 % / Rmerge(I) obs: 0.068 / Rsym value: 0.068 / Net I/σ(I): 14.661 |
| Reflection shell | Resolution: 1.5→1.53 Å / Redundancy: 3.6 % / Rmerge(I) obs: 0.319 / Mean I/σ(I) obs: 3.583 / Rsym value: 0.319 / % possible all: 92.8 |
| Cell measurement | Reflection used: 179388 |
-Phasing
| Phasing | Method:  molecular replacement molecular replacement | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR |
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT / Resolution: 1.5→19.504 Å / Occupancy max: 1 / Occupancy min: 0.41 / FOM work R set: 0.868 / SU ML: 0.15 / σ(F): 1.99 / Phase error: 20.54 / Stereochemistry target values: ML MOLECULAR REPLACEMENT / Resolution: 1.5→19.504 Å / Occupancy max: 1 / Occupancy min: 0.41 / FOM work R set: 0.868 / SU ML: 0.15 / σ(F): 1.99 / Phase error: 20.54 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 1 Å / VDW probe radii: 1.2 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 92.24 Å2 / Biso mean: 26.9222 Å2 / Biso min: 10.02 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.5→19.504 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 10
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj