| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| PHENIX | (PHENIX.REFINE)| 精密化 | | XDS | | データ削減 | | Aimless | | データスケーリング | | PHASER | | 位相決定 | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: PDB ENTRY 4C40
解像度: 2→37.415 Å / SU ML: 0.21 / σ(F): 1.35 / 位相誤差: 25.97 / 立体化学のターゲット値: ML
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.2344 | 225 | 4.6 % |
|---|
| Rwork | 0.1844 | - | - |
|---|
| obs | 0.1866 | 4916 | 98.32 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL |
|---|
| 原子変位パラメータ | Biso mean: 47.6 Å2 |
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 2→37.415 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 0 | 417 | 2 | 23 | 442 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.004 | 468 | | X-RAY DIFFRACTION | f_angle_d| 0.805 | 730 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 12.452 | 225 | | X-RAY DIFFRACTION | f_chiral_restr| 0.038 | 95 | | X-RAY DIFFRACTION | f_plane_restr| 0.007 | 19 | | | | | |
|
|---|
| LS精密化 シェル | | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Refine-ID | % reflection obs (%) |
|---|
| 2.0003-2.52 | 0.2906 | 119 | 0.2593 | 2280 | X-RAY DIFFRACTION | 99 | | 2.52-37.4212 | 0.2199 | 106 | 0.1687 | 2411 | X-RAY DIFFRACTION | 98 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 1.1672 | -1.4148 | -0.3635 | 2.4968 | 0.6478 | 0.1611 | 0.1663 | 0.3688 | -0.2482 | -0.5535 | 0.2658 | -0.543 | 0.8762 | 0.4257 | -0.3831 | 1.2678 | -0.043 | -0.0498 | 0.3025 | -0.1792 | 0.4911 | 91.7583 | -55.6521 | 100.5924 | | 2 | 4.3541 | 2.7042 | -1.6924 | 6.9953 | 3.4595 | 5.5886 | 0.3349 | -0.2724 | 0.3185 | 0.8342 | 0.021 | 0.05 | 1.2832 | -0.2298 | -0.2852 | 0.6174 | -0.168 | -0.0063 | 0.2838 | -0.0303 | 0.4378 | 87.9473 | -43.8944 | 105.332 | | 3 | 8.0021 | 2.1962 | 0.0615 | 9.043 | 2.4118 | 2.1142 | 0.4165 | 0.4286 | 0.4214 | 0.3222 | -0.3721 | 0.4439 | 0.0648 | -0.6301 | 0.0128 | 0.3838 | -0.0052 | 0.03 | 0.3434 | 0.0521 | 0.4109 | 87.2042 | -35.5507 | 91.3152 | | 4 | 8.2033 | -1.0688 | 1.7598 | 0.6572 | -2.135 | 7.4107 | 0.3643 | -0.0816 | 0.6847 | 0.4857 | 0.2248 | -0.3584 | -0.5832 | 2.3089 | 0.0912 | 0.2326 | 0.0461 | -0.0712 | 0.8648 | -0.0682 | 0.4221 | 103.0795 | -34.7043 | 92.491 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details |
|---|
| 1 | X-RAY DIFFRACTION | 1 | (CHAIN A AND RESID 1:4)| 2 | X-RAY DIFFRACTION | 2 | (CHAIN A AND RESID 5:8)| 3 | X-RAY DIFFRACTION | 3 | (CHAIN A AND RESID 9:13)| 4 | X-RAY DIFFRACTION | 4 | (CHAIN A AND RESID 14:19) | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HALOARCULA MARISMORTUI (好塩性)
HALOARCULA MARISMORTUI (好塩性) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Commun. / 年: 2014
ジャーナル: Nat.Commun. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4cs1.cif.gz
4cs1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4cs1.ent.gz
pdb4cs1.ent.gz PDB形式
PDB形式 4cs1.json.gz
4cs1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4cs1_validation.pdf.gz
4cs1_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4cs1_full_validation.pdf.gz
4cs1_full_validation.pdf.gz 4cs1_validation.xml.gz
4cs1_validation.xml.gz 4cs1_validation.cif.gz
4cs1_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/cs/4cs1
https://data.pdbj.org/pub/pdb/validation_reports/cs/4cs1 ftp://data.pdbj.org/pub/pdb/validation_reports/cs/4cs1
ftp://data.pdbj.org/pub/pdb/validation_reports/cs/4cs1
 リンク
リンク 集合体
集合体
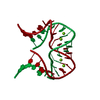
 要素
要素 HALOARCULA MARISMORTUI (好塩性)
HALOARCULA MARISMORTUI (好塩性) X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.9763
/ ビームライン: I03 / 波長: 0.9763  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー





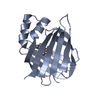





 PDBj
PDBj
































