Entry Database : PDB / ID : 3wznTitle Crystal structure of the core streptavidin mutant V21 (Y22S/N23D/S27D/Y83S/R84K/E101D/R103K/E116N) complexed with biotin at 1.3 A resolution Streptavidin Keywords / Function / homology Function Domain/homology Component
/ / / / / / / / / / / / / / / / / / / / Biological species Streptomyces avidinii (bacteria)Method / / / Resolution : 1.3 Å Authors Kawato, T. / Mizohata, E. / Shimizu, Y. / Meshizuka, T. / Yamamoto, T. / Takasu, N. / Matsuoka, M. / Matsumura, H. / Tsumoto, K. / Kodama, T. ...Kawato, T. / Mizohata, E. / Shimizu, Y. / Meshizuka, T. / Yamamoto, T. / Takasu, N. / Matsuoka, M. / Matsumura, H. / Tsumoto, K. / Kodama, T. / Kanai, M. / Doi, H. / Inoue, T. / Sugiyama, A. Journal : J.Biochem. / Year : 2015Title : Structure-based design of a streptavidin mutant specific for an artificial biotin analogue.Authors : Kawato, T. / Mizohata, E. / Shimizu, Y. / Meshizuka, T. / Yamamoto, T. / Takasu, N. / Matsuoka, M. / Matsumura, H. / Kodama, T. / Kanai, M. / Doi, H. / Inoue, T. / Sugiyama, A. History Deposition Oct 1, 2014 Deposition site / Processing site Revision 1.0 Feb 18, 2015 Provider / Type Revision 1.1 Nov 22, 2017 Group / Category / Item Revision 1.2 Aug 24, 2022 Group / Derived calculationsCategory citation / database_2 ... citation / database_2 / struct_ref_seq_dif / struct_site Item _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id Revision 1.3 Nov 8, 2023 Group / Refinement descriptionCategory / chem_comp_bond / pdbx_initial_refinement_model
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
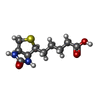
Components Keywords
Keywords Function and homology information
Function and homology information Streptomyces avidinii (bacteria)
Streptomyces avidinii (bacteria) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.3 Å
MOLECULAR REPLACEMENT / Resolution: 1.3 Å  Authors
Authors Citation
Citation Journal: J.Biochem. / Year: 2015
Journal: J.Biochem. / Year: 2015 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 3wzn.cif.gz
3wzn.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb3wzn.ent.gz
pdb3wzn.ent.gz PDB format
PDB format 3wzn.json.gz
3wzn.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads 3wzn_validation.pdf.gz
3wzn_validation.pdf.gz wwPDB validaton report
wwPDB validaton report 3wzn_full_validation.pdf.gz
3wzn_full_validation.pdf.gz 3wzn_validation.xml.gz
3wzn_validation.xml.gz 3wzn_validation.cif.gz
3wzn_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/wz/3wzn
https://data.pdbj.org/pub/pdb/validation_reports/wz/3wzn ftp://data.pdbj.org/pub/pdb/validation_reports/wz/3wzn
ftp://data.pdbj.org/pub/pdb/validation_reports/wz/3wzn



 Links
Links Assembly
Assembly

 Components
Components Streptomyces avidinii (bacteria) / Plasmid: pCold TF / Production host:
Streptomyces avidinii (bacteria) / Plasmid: pCold TF / Production host: 
 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  Photon Factory
Photon Factory  / Beamline: BL-17A / Wavelength: 0.98 Å
/ Beamline: BL-17A / Wavelength: 0.98 Å Processing
Processing MOLECULAR REPLACEMENT
MOLECULAR REPLACEMENT Movie
Movie Controller
Controller












 PDBj
PDBj