+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3bwn | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | L-tryptophan aminotransferase | ||||||
 要素 要素 | (L-tryptophan ...) x 2 | ||||||
 キーワード キーワード | TRANSFERASE / aminotransferase / auxin synthesis / tryptophan / pyridoxal-5'-phosphate / indole-3-pyruvate | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tryptophan transaminase / L-tryptophan-pyruvate aminotransferase / indoleacetic acid biosynthetic process / cotyledon vascular tissue pattern formation / L-phenylalanine-pyruvate transaminase activity / L-tryptophan:2-oxoglutarate aminotransferase activity / L-tryptophan:pyruvate aminotransferase activity / L-tyrosine-pyruvate transaminase activity / L-methionine:2-oxoglutarate aminotransferase activity / gynoecium development ...tryptophan transaminase / L-tryptophan-pyruvate aminotransferase / indoleacetic acid biosynthetic process / cotyledon vascular tissue pattern formation / L-phenylalanine-pyruvate transaminase activity / L-tryptophan:2-oxoglutarate aminotransferase activity / L-tryptophan:pyruvate aminotransferase activity / L-tyrosine-pyruvate transaminase activity / L-methionine:2-oxoglutarate aminotransferase activity / gynoecium development / L-phenylalanine-2-oxoglutarate transaminase activity / L-alanine:2-oxoglutarate aminotransferase activity / primary root development / maintenance of root meristem identity / cotyledon development / shoot system development / shade avoidance / carbon-sulfur lyase activity / phloem or xylem histogenesis / response to ethylene / positive gravitropism / leaf development / flower development / auxin polar transport / root development / L-tyrosine-2-oxoglutarate transaminase activity / embryo development ending in seed dormancy / pyridoxal phosphate binding / defense response to bacterium / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / RIGID BODY / 解像度: 2.25 Å シンクロトロン / RIGID BODY / 解像度: 2.25 Å | ||||||
 データ登録者 データ登録者 | Ferrer, J.-L. / Noel, J.P. / Pojer, F. / Bowman, M. / Chory, J. / Tao, Y. | ||||||
 引用 引用 |  ジャーナル: Cell(Cambridge,Mass.) / 年: 2008 ジャーナル: Cell(Cambridge,Mass.) / 年: 2008タイトル: Rapid synthesis of auxin via a new tryptophan-dependent pathway is required for shade avoidance in plants 著者: Tao, Y. / Ferrer, J.L. / Ljung, K. / Pojer, F. / Hong, F. / Long, J.A. / Li, L. / Moreno, J.E. / Bowman, M.E. / Ivans, L.J. / Cheng, Y. / Lim, J. / Zhao, Y. / Ballare, C.L. / Sandberg, G. / Noel, J.P. / Chory, J. #1:  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Structural basis for entry of L-TRP into pathway for auxin biosynthesis 著者: Ferrer, J.-L. / Tao, Y. / Pojer, F. / Bowman, M.E. / Chory, J. / Noel, J.P. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3bwn.cif.gz 3bwn.cif.gz | 861.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3bwn.ent.gz pdb3bwn.ent.gz | 710.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3bwn.json.gz 3bwn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3bwn_validation.pdf.gz 3bwn_validation.pdf.gz | 546.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3bwn_full_validation.pdf.gz 3bwn_full_validation.pdf.gz | 611.4 KB | 表示 | |
| XML形式データ |  3bwn_validation.xml.gz 3bwn_validation.xml.gz | 90.3 KB | 表示 | |
| CIF形式データ |  3bwn_validation.cif.gz 3bwn_validation.cif.gz | 123 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bw/3bwn https://data.pdbj.org/pub/pdb/validation_reports/bw/3bwn ftp://data.pdbj.org/pub/pdb/validation_reports/bw/3bwn ftp://data.pdbj.org/pub/pdb/validation_reports/bw/3bwn | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1lk9S S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-L-tryptophan ... , 2種, 6分子 ABDEFC
| #1: タンパク質 | 分子量: 44864.020 Da / 分子数: 5 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: At1g70560/F5A18_26, F5A18.26 / 発現宿主:  #2: タンパク質 | | 分子量: 45092.133 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: At1g70560/F5A18_26, F5A18.26 / 発現宿主:  |
|---|
-非ポリマー , 4種, 598分子 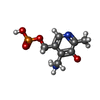






| #3: 化合物 | ChemComp-PMP / #4: 化合物 | ChemComp-PO4 / | #5: 化合物 | ChemComp-PHE / | #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.29 Å3/Da / 溶媒含有率: 46.32 % |
|---|---|
| 結晶化 | 温度: 293 K / pH: 7 詳細: 13-19% PEG 3350, 0.3M KCl, 100mM MOPSO pH7.0, VAPOR DIFFUSION, HANGING DROP, temperature 293.0K, pH 7.00 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL1-5 / 波長: 0.97944 / ビームライン: BL1-5 / 波長: 0.97944 |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2006年3月4日 / 詳細: MIRRORS + DOUBLE MONOCHROMATOR |
| 放射 | モノクロメーター: SI (111) DOUBLE MONOCHROMATOR / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.97944 Å / 相対比: 1 |
| 反射 | 解像度: 2.25→30 Å / Num. obs: 110711 / % possible obs: 90 % / Observed criterion σ(I): 0 / 冗長度: 2.49 % / Rsym value: 0.56 / Net I/σ(I): 12.5 |
| 反射 シェル | 解像度: 2.25→2.31 Å / 冗長度: 2.41 % / Mean I/σ(I) obs: 2.53 / Rsym value: 0.414 / % possible all: 82.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: RIGID BODY 開始モデル: PDB ENTRY 1LK9 解像度: 2.25→29.66 Å / Cor.coef. Fo:Fc: 0.947 / Cor.coef. Fo:Fc free: 0.93 / SU B: 19.051 / SU ML: 0.203 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.244 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 46.99 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.25→29.66 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.25→2.31 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





