+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3bvd | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of Surface-engineered Cytochrome ba3 Oxidase from Thermus thermophilus under Xenon Pressure, 100psi 5min | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | OXIDOREDUCTASE / CYTOCHROME BA3 OXIDASE / HEME / INTEGRAL MEMBRANE PROTEIN / COPPER / ELECTRON TRANSPORT / HYDROGEN ION TRANSPORT / ION TRANSPORT / IRON / METAL-BINDING / RESPIRATORY CHAIN / TRANSMEMBRANE / TRANSPORT / FORMYLATION / XENON | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytochrome-c oxidase / oxidative phosphorylation / cytochrome-c oxidase activity / copper ion binding / heme binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.37 Å 分子置換 / 解像度: 3.37 Å | ||||||
 データ登録者 データ登録者 | Luna, V.M. / Chen, Y. / Fee, J.A. / Stout, C.D. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2008 ジャーナル: Biochemistry / 年: 2008タイトル: Crystallographic Studies of Xe and Kr Binding within the Large Internal Cavity of Cytochrome ba3 from Thermus thermophilus: Structural Analysis and Role of Oxygen Transport Channels in the Heme-Cu Oxidases. 著者: Luna, V.M. / Chen, Y. / Fee, J.A. / Stout, C.D. #1:  ジャーナル: Acta Crystallogr.,Sect.F / 年: 2007 ジャーナル: Acta Crystallogr.,Sect.F / 年: 2007タイトル: An unexpected outcome of surface engineering an integral membrane protein: improved crystallization of cytochrome ba3 from Thermus thermophilus 著者: Liu, B. / Luna, V.M. / Chen, Y. / Stout, C.D. / Fee, J.A. #2:  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2005タイトル: A novel cryoprotection scheme for enchancing the diffraction of crystals of recombinant cytochrome ba3 oxidase from Thermus thermophilus 著者: Hunsicker-Wang, L.M. / Pacoma, R.L. / Chen, Y. / Fee, J.A. / Stout, C.D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3bvd.cif.gz 3bvd.cif.gz | 146.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3bvd.ent.gz pdb3bvd.ent.gz | 109.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3bvd.json.gz 3bvd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3bvd_validation.pdf.gz 3bvd_validation.pdf.gz | 940.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3bvd_full_validation.pdf.gz 3bvd_full_validation.pdf.gz | 961.4 KB | 表示 | |
| XML形式データ |  3bvd_validation.xml.gz 3bvd_validation.xml.gz | 26.9 KB | 表示 | |
| CIF形式データ |  3bvd_validation.cif.gz 3bvd_validation.cif.gz | 36.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bv/3bvd https://data.pdbj.org/pub/pdb/validation_reports/bv/3bvd ftp://data.pdbj.org/pub/pdb/validation_reports/bv/3bvd ftp://data.pdbj.org/pub/pdb/validation_reports/bv/3bvd | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1xmeS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Cytochrome c oxidase subunit ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 63430.008 Da / 分子数: 1 / 変異: K258R / 由来タイプ: 組換発現 由来: (組換発現)   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア)株: HB8 / 遺伝子: cbaA / プラスミド: PMK18 / 発現宿主:   Thermus thermophilus (バクテリア) / 株 (発現宿主): HB8 / 参照: UniProt: Q5SJ79, cytochrome-c oxidase Thermus thermophilus (バクテリア) / 株 (発現宿主): HB8 / 参照: UniProt: Q5SJ79, cytochrome-c oxidase |
|---|---|
| #2: タンパク質 | 分子量: 18580.314 Da / 分子数: 1 / 変異: E4Q / 由来タイプ: 組換発現 由来: (組換発現)   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア)株: HB8 / 遺伝子: cbaB, ctaC / プラスミド: PMK18 / 発現宿主:   Thermus thermophilus (バクテリア) / 株 (発現宿主): HB8 / 参照: UniProt: Q5SJ80, cytochrome-c oxidase Thermus thermophilus (バクテリア) / 株 (発現宿主): HB8 / 参照: UniProt: Q5SJ80, cytochrome-c oxidase |
-タンパク質・ペプチド , 1種, 1分子 C
| #3: タンパク質・ペプチド | 分子量: 3769.602 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア)株: HB8 / 遺伝子: cbaD / プラスミド: PMK18 / 発現宿主:   Thermus thermophilus (バクテリア) / 株 (発現宿主): HB8 / 参照: UniProt: P82543, cytochrome-c oxidase Thermus thermophilus (バクテリア) / 株 (発現宿主): HB8 / 参照: UniProt: P82543, cytochrome-c oxidase |
|---|
-非ポリマー , 5種, 11分子 

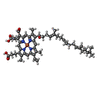






| #4: 化合物 | ChemComp-CU / | ||
|---|---|---|---|
| #5: 化合物 | ChemComp-HEM / | ||
| #6: 化合物 | ChemComp-HAS / | ||
| #7: 化合物 | ChemComp-XE / #8: 化合物 | ChemComp-CUA / | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.21 Å3/Da / 溶媒含有率: 61.65 % |
|---|---|
| 結晶化 | 温度: 297 K / 手法: 蒸気拡散法 / pH: 7 詳細: 6-9% PEG 2000, 50mM KCl, 7.5-30mM bis-Tris pH 7.0, 6.5mM nonyl-B-D-glucopyranoside , VAPOR DIFFUSION, temperature 297K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL9-1 / 波長: 0.979 Å / ビームライン: BL9-1 / 波長: 0.979 Å |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2007年2月2日 詳細: Vertical focusing mirror; single crystal Si(311) bent monochromator (horizontal focusing) |
| 放射 | モノクロメーター: Side-scattering cuberoot I-beam bent single crystal; asymmetric cut 12.2 degs. プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.979 Å / 相対比: 1 |
| 反射 | 解像度: 3.37→27.16 Å / Num. all: 16390 / Num. obs: 16341 / % possible obs: 99.7 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 4.5 % / Biso Wilson estimate: 108.222 Å2 / Rmerge(I) obs: 0.068 / Net I/σ(I): 8.1 |
| 反射 シェル | 解像度: 3.37→3.55 Å / 冗長度: 4.8 % / Rmerge(I) obs: 0.463 / Mean I/σ(I) obs: 1.8 / Num. unique all: 2339 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1XME 解像度: 3.37→26.96 Å / Cor.coef. Fo:Fc: 0.891 / Cor.coef. Fo:Fc free: 0.826 / SU B: 38.25 / SU ML: 0.624 / Isotropic thermal model: Overall / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.712 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 85.48 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.37→26.96 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 3.37→3.58 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj







