+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2xjh | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
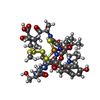
| タイトル | Structure and Copper-binding Properties of Methanobactins from Methylosinus trichosporium OB3b | |||||||||
 要素 要素 | METHANOBACTIN MB-OB3B | |||||||||
 キーワード キーワード | OXIDOREDUCTASE / METAL TRANSPORT / METHANOTROPHS / OXAZALONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報copper ion transport / superoxide dismutase / superoxide dismutase activity / 酸化還元酵素; 過酸化物を電子受容体にする; ペルオキシダーゼ / peroxidase activity / extracellular region / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  METHYLOSINUS TRICHOSPORIUM (バクテリア) METHYLOSINUS TRICHOSPORIUM (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 0.92 Å 単波長異常分散 / 解像度: 0.92 Å | |||||||||
 データ登録者 データ登録者 | El-Ghazouani, A. / Basle, A. / Firbank, S.J. / Knapp, C.W. / Gray, J. / Graham, D.W. / Dennison, C. | |||||||||
 引用 引用 |  ジャーナル: Inorg.Chem. / 年: 2011 ジャーナル: Inorg.Chem. / 年: 2011タイトル: Copper-Binding Properties and Structures of Methanobactins from Methylosinus Trichosporium Ob3B. 著者: El Ghazouani, A. / Basle, A. / Firbank, S.J. / Knapp, C.W. / Gray, J. / Graham, D.W. / Dennison, C. #1: ジャーナル: Science / 年: 2004 タイトル: Methanobactin, a Copper-Acquisition Compound from Methane-Oxidizing Bacteria. 著者: Kim, H.J. / Graham, D.W. / Dispirito, A.A. / Alterman, M.A. / Galeva, N. / Larive, C.K. / Asunskis, D. / Sherwood, P.M.A. #2: ジャーナル: J.Am.Chem.Soc. / 年: 2008 タイトル: NMR, Mass Spectrometry and Chemical Evidence Reveal a Different Chemical Structure for Methanobactin that Contains Oxazolone Rings. 著者: Behling, L.A. / Hartsel, S.C. / Lewis, D.E. / Dispirito, A.A. / Choi, D.W. / Masterson, L.R. / Veglia, G. / Gallagher, W.H. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2xjh.cif.gz 2xjh.cif.gz | 21.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2xjh.ent.gz pdb2xjh.ent.gz | 14.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2xjh.json.gz 2xjh.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2xjh_validation.pdf.gz 2xjh_validation.pdf.gz | 425.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2xjh_full_validation.pdf.gz 2xjh_full_validation.pdf.gz | 425.5 KB | 表示 | |
| XML形式データ |  2xjh_validation.xml.gz 2xjh_validation.xml.gz | 3.8 KB | 表示 | |
| CIF形式データ |  2xjh_validation.cif.gz 2xjh_validation.cif.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xj/2xjh https://data.pdbj.org/pub/pdb/validation_reports/xj/2xjh ftp://data.pdbj.org/pub/pdb/validation_reports/xj/2xjh ftp://data.pdbj.org/pub/pdb/validation_reports/xj/2xjh | HTTPS FTP |
-関連構造データ
| 関連構造データ |  2xjiC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||
| 2 | 
| |||||||||
| 単位格子 |
| |||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質・ペプチド | | #2: 化合物 | #3: 化合物 | #4: 水 | ChemComp-HOH / | 構成要素の詳細 | METHANOBACTIN IS A MEMBER OF THE FAMILY OF CHALKOPHORES, A COPPER CHELATING AGENT INVOLVED IN ...METHANOBAC | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.41 Å3/Da / 溶媒含有率: 13 % 解説: PREVIOUSLY SOLVED MODEL CCDC BIDLOQ WAS MANUALLY FITTED INTO DENSITY |
|---|---|
| 結晶化 | pH: 7.5 / 詳細: 4M SODIUM FORMATE, pH 7.5 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I04 / 波長: 0.91 / ビームライン: I04 / 波長: 0.91 |
| 検出器 | タイプ: ADSC CCD / 検出器: CCD / 日付: 2009年8月1日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.91 Å / 相対比: 1 |
| 反射 | 解像度: 0.92→20.84 Å / Num. obs: 9025 / % possible obs: 95.7 % / Observed criterion σ(I): 2 / 冗長度: 11.3 % / Rmerge(I) obs: 0.06 / Net I/σ(I): 25.1 |
| 反射 シェル | 解像度: 0.92→0.97 Å / 冗長度: 5.5 % / Rmerge(I) obs: 0.34 / Mean I/σ(I) obs: 5.5 / % possible all: 78.8 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  単波長異常分散 単波長異常分散開始モデル: NONE 解像度: 0.92→20 Å / Num. parameters: 1623 / Num. restraintsaints: 1871 / 交差検証法: FREE R-VALUE / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER
| |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 13 / Occupancy sum hydrogen: 92 / Occupancy sum non hydrogen: 165.46 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 0.92→20 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj