+ データを開く
データを開く
- 基本情報
基本情報
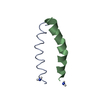
| 登録情報 | データベース: PDB / ID: 2u2a | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
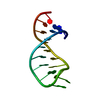
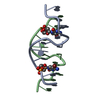
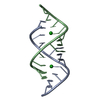
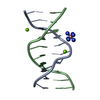
| タイトル | STEM LOOP IIA FROM U2 SNRNA OF SACCHAROMYCES CEREVISIAE, NMR, MINIMIZED AVERAGE STRUCTURE | ||||||||||||||||||||
 要素 要素 | RNA (5'-R(P* キーワード キーワードRNA / PRE-MRNA SPLICING / U2SNRNA / U-TURN / RNA-PROTEIN INTERACTIONS / RIBONUCLEIC ACID | 機能・相同性 | RNA / RNA (> 10) |  機能・相同性情報 機能・相同性情報生物種 |  手法 | 溶液NMR / molecular dynamics |  データ登録者 データ登録者Stallings, S.C. / Moore, P.B. |  引用 引用 ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: The structure of an essential splicing element: stem loop IIa from yeast U2 snRNA. 著者: Stallings, S.C. / Moore, P.B. #1:  ジャーナル: Genes Dev. / 年: 1991 ジャーナル: Genes Dev. / 年: 1991タイトル: Efficient Association of U2 Snrnps with Pre-Mrna Requires an Essential U2 RNA Structural Element 著者: Zavanelli, M.I. / Ares Junior, M. #2:  ジャーナル: Genes Dev. / 年: 1990 ジャーナル: Genes Dev. / 年: 1990タイトル: Lethal and Temperature-Sensitive Mutations and Their Suppressors Identify an Essential Structural Element in U2 Small Nuclear RNA 著者: Ares Junior, M. / Igel, A.H. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2u2a.cif.gz 2u2a.cif.gz | 20.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2u2a.ent.gz pdb2u2a.ent.gz | 13.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2u2a.json.gz 2u2a.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2u2a_validation.pdf.gz 2u2a_validation.pdf.gz | 291.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2u2a_full_validation.pdf.gz 2u2a_full_validation.pdf.gz | 291.5 KB | 表示 | |
| XML形式データ |  2u2a_validation.xml.gz 2u2a_validation.xml.gz | 1.6 KB | 表示 | |
| CIF形式データ |  2u2a_validation.cif.gz 2u2a_validation.cif.gz | 1.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/u2/2u2a https://data.pdbj.org/pub/pdb/validation_reports/u2/2u2a ftp://data.pdbj.org/pub/pdb/validation_reports/u2/2u2a ftp://data.pdbj.org/pub/pdb/validation_reports/u2/2u2a | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 6406.878 Da / 分子数: 1 / 断片: STEM LOOP IIA 変異: TWO TERMINAL BASE PAIRS WERE CHANGED FROM UAS IN STEM LOOP IIA TO GCS IN U2A TO ACCOMMODATE T7 TRANSCRIPTION 由来タイプ: 組換発現 由来: (組換発現)  解説: MOLECULE TRANSCRIBED FROM A PARTIALLY SINGLE-STRANDED DNA TEMPLATE USING T7 RNA POLYMERASE 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 試料状態 | イオン強度: 75 mM / pH: 7.6 圧: 1 ATM SOLVENT SYSTEM : 10MM KH2PO4, 50 MM KCL, 15 MM NACL, 0.5 MM EDTA atm 温度: 303 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR software |
| ||||||||||||||||
| 精密化 | 手法: molecular dynamics / ソフトェア番号: 1 詳細: REFINEMENT WAS DONE USING TORSION ANGLE MOLECULAR DYNAMICS. DETAILS OF THE PROTOCOL CAN BE FOUND IN THE STRUCTURE CITATION ABOVE. | ||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATIONS 計算したコンフォーマーの数: 15 / 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj





























 HSQC
HSQC