+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2otb | ||||||
|---|---|---|---|---|---|---|---|
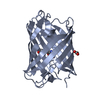
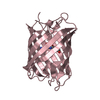
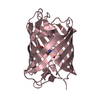
| タイトル | Crystal structure of a monomeric cyan fluorescent protein in the fluorescent state | ||||||
 要素 要素 | GFP-like fluorescent chromoprotein cFP484 | ||||||
 キーワード キーワード | FLUORESCENT PROTEIN / Beta can | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Clavularia sp. (無脊椎動物) Clavularia sp. (無脊椎動物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.79 Å 分子置換 / 解像度: 1.79 Å | ||||||
 データ登録者 データ登録者 | Henderson, J.N. / Ai, H. / Campbell, R.E. / Remington, S.J. | ||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007タイトル: Structural basis for reversible photobleaching of a green fluorescent protein homologue. 著者: Henderson, J.N. / Ai, H.W. / Campbell, R.E. / Remington, S.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2otb.cif.gz 2otb.cif.gz | 108.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2otb.ent.gz pdb2otb.ent.gz | 81.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2otb.json.gz 2otb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2otb_validation.pdf.gz 2otb_validation.pdf.gz | 426.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2otb_full_validation.pdf.gz 2otb_full_validation.pdf.gz | 430.7 KB | 表示 | |
| XML形式データ |  2otb_validation.xml.gz 2otb_validation.xml.gz | 22.5 KB | 表示 | |
| CIF形式データ |  2otb_validation.cif.gz 2otb_validation.cif.gz | 33.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ot/2otb https://data.pdbj.org/pub/pdb/validation_reports/ot/2otb ftp://data.pdbj.org/pub/pdb/validation_reports/ot/2otb ftp://data.pdbj.org/pub/pdb/validation_reports/ot/2otb | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | The biological assembly is a monomer. There are two monomers in the asymmetric unit. |
- 要素
要素
| #1: タンパク質 | 分子量: 24799.186 Da / 分子数: 2 変異: H42N, L44I, S62T, Q66A, L72F, A80P, D81N, R123H, F124L, D125K, M127E, M150L, S162K, S164K, Y173H, C175V, S179T, K182R, V186A, L213V, N216S 由来タイプ: 組換発現 / 由来: (組換発現)  Clavularia sp. (無脊椎動物) / 遺伝子: cFP484 / プラスミド: pBAD/HisB / 発現宿主: Clavularia sp. (無脊椎動物) / 遺伝子: cFP484 / プラスミド: pBAD/HisB / 発現宿主:  #2: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.12 Å3/Da / 溶媒含有率: 41.88 % |
|---|---|
| 結晶化 | 温度: 295 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.8 詳細: 31% PEG 3400, 0.2 M Lithium Sulfate, 0.1 M Sodium Acetate, 2.9 mM 1-s-nonyl-beta-D-thioglucoside, pH 4.8, VAPOR DIFFUSION, HANGING DROP, temperature 295K |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 5.0.2 / 波長: 1 / ビームライン: 5.0.2 / 波長: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2005年12月5日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: Double-crystal, Si(111) liquid N2 cooled プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 1 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | 冗長度: 6.8 % / Av σ(I) over netI: 14.8 / 数: 273631 / Rmerge(I) obs: 0.073 / Χ2: 1.05 / D res high: 1.79 Å / D res low: 50 Å / Num. obs: 40185 / % possible obs: 99.4 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Diffraction reflection shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.79→50 Å / Num. obs: 40185 / % possible obs: 99.4 % / Observed criterion σ(F): -3 / Observed criterion σ(I): -3 / 冗長度: 6.8 % / Rmerge(I) obs: 0.073 / Χ2: 1.049 / Net I/σ(I): 14.8 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | 解像度: 1.79→1.85 Å / 冗長度: 6.6 % / Rmerge(I) obs: 0.176 / Mean I/σ(I) obs: 21.1 / Num. unique all: 3953 / Χ2: 1.056 / % possible all: 98.9 |
-位相決定
| Phasing MR | Rfactor: 0.669 / Cor.coef. Fo:Fc: 0.245
|
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB Entry 1ZUX, chain A 解像度: 1.79→50 Å / Isotropic thermal model: Isotropic / 交差検証法: THROUGHOUT / σ(F): 2 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | Bsol: 150 Å2 / ksol: 0.8 e/Å3 | ||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 18.622 Å2
| ||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.79→50 Å
| ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.79→1.85 Å / Num. reflection obs: 3953 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj


