+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2mfj | ||||||
|---|---|---|---|---|---|---|---|
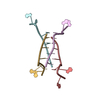
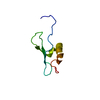
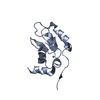
| タイトル | Solution structure of Blo t 19, a minor dust mite allergen from Blomia tropicalis | ||||||
 要素 要素 | Blo t 19 | ||||||
 キーワード キーワード | ALLERGEN / Anti-microbial Protein | ||||||
| 機能・相同性 | Antibacterial factor-related peptide / Antibacterial factor-related peptide superfamily / Nematode antimicrobial peptide / defense response to other organism / Blo t 19 allergen 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Blomia tropicalis (ダニ) Blomia tropicalis (ダニ) | ||||||
| 手法 | 溶液NMR / torsion angle dynamics, DGSA-distance geometry simulated annealing | ||||||
| Model details | lowest energy, model1 | ||||||
 データ登録者 データ登録者 | Naik, M.T. / Naik, N. / Huang, T. | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Solution structure of Blo 1 19 著者: Naik, M. / Naik, N. / Kuo, I. / Liao, Y. / Huang, T. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2mfj.cif.gz 2mfj.cif.gz | 363.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2mfj.ent.gz pdb2mfj.ent.gz | 303.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2mfj.json.gz 2mfj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2mfj_validation.pdf.gz 2mfj_validation.pdf.gz | 542.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2mfj_full_validation.pdf.gz 2mfj_full_validation.pdf.gz | 730.6 KB | 表示 | |
| XML形式データ |  2mfj_validation.xml.gz 2mfj_validation.xml.gz | 35.7 KB | 表示 | |
| CIF形式データ |  2mfj_validation.cif.gz 2mfj_validation.cif.gz | 51.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mf/2mfj https://data.pdbj.org/pub/pdb/validation_reports/mf/2mfj ftp://data.pdbj.org/pub/pdb/validation_reports/mf/2mfj ftp://data.pdbj.org/pub/pdb/validation_reports/mf/2mfj | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6945.833 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Blomia tropicalis (ダニ) / 発現宿主: Blomia tropicalis (ダニ) / 発現宿主:  |
|---|---|
| Has protein modification | Y |
| 配列の詳細 | FIRST TWO RESIDUES (GS) ARE EXPRESSION |
-実験情報
-実験
| 実験 | 手法: 溶液NMR 詳細: The data from this entry were used to determine structure of Blo t 19. Due to Proline isomerization, Blo t 19 spectrum has degenerate resonances for a stretch of six residues in its C- ...詳細: The data from this entry were used to determine structure of Blo t 19. Due to Proline isomerization, Blo t 19 spectrum has degenerate resonances for a stretch of six residues in its C-terminus. These two populations have varying intensities and are unambiguously assigned as major and minor population. Solution structure is calculated for the Major population. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: NMR data was acquired at 295K using Shigemi NMR tubes. |
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 試料状態 | pH: 6 / 圧: ambient / 温度: 295 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: torsion angle dynamics, DGSA-distance geometry simulated annealing ソフトェア番号: 1 詳細: Initial structure ensemble was calculated by semi-automated NOESY assignment by CYANA. The assignments were manually verified in Sparky and final structure annealing was performed in CYANA. ...詳細: Initial structure ensemble was calculated by semi-automated NOESY assignment by CYANA. The assignments were manually verified in Sparky and final structure annealing was performed in CYANA. Structure and restraints from CYANA were imported in Xplor-NIH for explicit water refinement. | ||||||||||||||||||||||||||||||||||||||||
| NMR constraints | NOE constraints total: 1140 / NOE intraresidue total count: 212 / NOE long range total count: 294 / NOE medium range total count: 314 / NOE sequential total count: 320 / Disulfide bond constraints total count: 12 / Hydrogen bond constraints total count: 54 / Protein chi angle constraints total count: 0 / Protein other angle constraints total count: 43 / Protein phi angle constraints total count: 39 / Protein psi angle constraints total count: 39 | ||||||||||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||||||||||||||
| NMRアンサンブル | Average torsion angle constraint violation: 0.08688 ° コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 400 / 登録したコンフォーマーの数: 20 / Maximum torsion angle constraint violation: 4.9 ° / Maximum upper distance constraint violation: 0.7 Å / 代表コンフォーマー: 1 / Torsion angle constraint violation method: PSVS | ||||||||||||||||||||||||||||||||||||||||
| NMR ensemble rms | Distance rms dev: 0.02648 Å / Distance rms dev error: 0.02581 Å |
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj HSQC
HSQC