+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2lzw | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
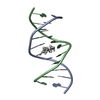
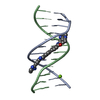
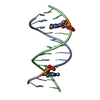
| タイトル | DNA duplex containing mispair-aligned O6G-heptylene-O6G interstrand cross-link | ||||||||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / INTERSTRAND CROSS-LINK | 機能・相同性 | HEPTANE / DNA / DNA (> 10) |  機能・相同性情報 機能・相同性情報生物種 | synthetic construct (人工物) | 手法 | 溶液NMR / simulated annealing | Model details | minimized average structure, model 1 | Model type details | minimized average |  データ登録者 データ登録者Denisov, A.Y. / McManus, F.P. / Noronha, A.M. / Wilds, C.J. |  引用 引用 ジャーナル: Org.Biomol.Chem. / 年: 2017 ジャーナル: Org.Biomol.Chem. / 年: 2017タイトル: Structural basis of interstrand cross-link repair by O 6 -alkylguanine DNA alkyltransferase. 著者: Denisov, A.Y. / McManus, F.P. / O'Flaherty, D.K. / Noronha, A.M. / Wilds, C.J. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2lzw.cif.gz 2lzw.cif.gz | 169.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2lzw.ent.gz pdb2lzw.ent.gz | 139.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2lzw.json.gz 2lzw.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lz/2lzw https://data.pdbj.org/pub/pdb/validation_reports/lz/2lzw ftp://data.pdbj.org/pub/pdb/validation_reports/lz/2lzw ftp://data.pdbj.org/pub/pdb/validation_reports/lz/2lzw | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
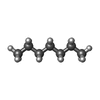
| #1: DNA鎖 | 分子量: 3373.222 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) #2: 化合物 | ChemComp-HP6 / | |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| |||||||||||||||
| 試料状態 |
|
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 | ||||||||||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations and the lowest energy 計算したコンフォーマーの数: 20 / 登録したコンフォーマーの数: 11 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj







































 HSQC
HSQC