+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2k71 | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure and dynamics of a DNA GNRA hairpin solved vy high-sensitivity NMR with two independent converging methods, simulated annealing (DYANA) and mesoscopic molecular modelling (BCE/AMBER) | ||||||||||||||||||||
 要素 要素 | 5'-D(* キーワード キーワードDNA / tetraloop hairpin / B-DNA-like / 2 non canonical torsions / unusual BIIz+ torsions | 機能・相同性 | DNA |  機能・相同性情報 機能・相同性情報手法 | 溶液NMR / simulated annealing, Biopolymer Chain Elasticity approach | Model details | DNA GNRA tetraloop structure |  データ登録者 データ登録者Santini, G.P.H. / Cognet, J.A.H. / Xu, D. / Singarapu, K.K. / Herve du Penhoat, C.L.M. |  引用 引用 ジャーナル: J.Phys.Chem.B / 年: 2009 ジャーナル: J.Phys.Chem.B / 年: 2009タイトル: Nucleic acid folding determined by mesoscale modeling and NMR spectroscopy: solution structure of d(GCGAAAGC). 著者: Santini, G.P. / Cognet, J.A. / Xu, D. / Singarapu, K.K. / Herve du Penhoat, C. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2k71.cif.gz 2k71.cif.gz | 89.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2k71.ent.gz pdb2k71.ent.gz | 60.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2k71.json.gz 2k71.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/k7/2k71 https://data.pdbj.org/pub/pdb/validation_reports/k7/2k71 ftp://data.pdbj.org/pub/pdb/validation_reports/k7/2k71 ftp://data.pdbj.org/pub/pdb/validation_reports/k7/2k71 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 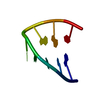
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 2460.644 Da / 分子数: 1 / 断片: DNA GNRA hairpin / 由来タイプ: 合成 |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR / 詳細: DNA GNRA tetraloop structure | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| |||||||||||||||||||||||||||||||||
| 試料状態 |
|
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing, Biopolymer Chain Elasticity approach ソフトェア番号: 1 詳細: 100 structures were used in DYANA simulated annealing approach to give models 1 to 20 that were energy-refined with AMBER, Model number 21 was constructed with the BCE approach in agreement ...詳細: 100 structures were used in DYANA simulated annealing approach to give models 1 to 20 that were energy-refined with AMBER, Model number 21 was constructed with the BCE approach in agreement with the NMR data and then energy-refined with AMBER | ||||||||||||||||||
| 代表構造 | 選択基準: fewest violations | ||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations 計算したコンフォーマーの数: 101 / 登録したコンフォーマーの数: 21 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj

 HSQC
HSQC