+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2gk2 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of the N terminal domain of human CEACAM1 | ||||||
 要素 要素 | Carcinoembryonic antigen-related cell adhesion molecule 1 | ||||||
 キーワード キーワード | CELL ADHESION / immunoglobulin domain / adhesion protein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of endothelial cell differentiation / insulin receptor internalization / negative regulation of cytotoxic T cell degranulation / Regulation of MITF-M dependent genes involved in invasion / granulocyte colony-stimulating factor signaling pathway / regulation of homophilic cell adhesion / negative regulation of hepatocyte proliferation / filamin binding / regulation of epidermal growth factor receptor signaling pathway / regulation of blood vessel remodeling ...regulation of endothelial cell differentiation / insulin receptor internalization / negative regulation of cytotoxic T cell degranulation / Regulation of MITF-M dependent genes involved in invasion / granulocyte colony-stimulating factor signaling pathway / regulation of homophilic cell adhesion / negative regulation of hepatocyte proliferation / filamin binding / regulation of epidermal growth factor receptor signaling pathway / regulation of blood vessel remodeling / regulation of sprouting angiogenesis / negative regulation of lipid biosynthetic process / negative regulation of natural killer cell mediated cytotoxicity directed against tumor cell target / negative regulation of T cell mediated cytotoxicity / regulation of endothelial cell migration / negative regulation of granulocyte differentiation / regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / insulin catabolic process / Fibronectin matrix formation / common myeloid progenitor cell proliferation / cell-cell adhesion via plasma-membrane adhesion molecules / negative regulation of interleukin-1 production / negative regulation of fatty acid biosynthetic process / positive regulation of vasculogenesis / negative regulation of platelet aggregation / bile acid transmembrane transporter activity / negative regulation of vascular permeability / regulation of immune system process / wound healing, spreading of cells / negative regulation of T cell receptor signaling pathway / transport vesicle membrane / bile acid and bile salt transport / microvillus membrane / blood vessel development / homophilic cell adhesion via plasma membrane adhesion molecules / tertiary granule membrane / lateral plasma membrane / regulation of ERK1 and ERK2 cascade / specific granule membrane / negative regulation of protein kinase activity / protein tyrosine kinase binding / basal plasma membrane / regulation of cell migration / integrin-mediated signaling pathway / Cell surface interactions at the vascular wall / adherens junction / regulation of cell growth / kinase binding / cellular response to insulin stimulus / cell-cell junction / cell junction / cell migration / actin binding / angiogenesis / protein phosphatase binding / calmodulin binding / protein dimerization activity / cell adhesion / apical plasma membrane / Neutrophil degranulation / cell surface / signal transduction / protein homodimerization activity / extracellular exosome / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Fedarovich, A. / Tomberg, J. / Nicholas, R.A. / Davies, C. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2006 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2006タイトル: Structure of the N-terminal domain of human CEACAM1: binding target of the opacity proteins during invasion of Neisseria meningitidis and N. gonorrhoeae. 著者: Fedarovich, A. / Tomberg, J. / Nicholas, R.A. / Davies, C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2gk2.cif.gz 2gk2.cif.gz | 58.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2gk2.ent.gz pdb2gk2.ent.gz | 42 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2gk2.json.gz 2gk2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2gk2_validation.pdf.gz 2gk2_validation.pdf.gz | 448.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2gk2_full_validation.pdf.gz 2gk2_full_validation.pdf.gz | 454.9 KB | 表示 | |
| XML形式データ |  2gk2_validation.xml.gz 2gk2_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  2gk2_validation.cif.gz 2gk2_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gk/2gk2 https://data.pdbj.org/pub/pdb/validation_reports/gk/2gk2 ftp://data.pdbj.org/pub/pdb/validation_reports/gk/2gk2 ftp://data.pdbj.org/pub/pdb/validation_reports/gk/2gk2 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1l6zS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 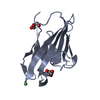
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 12171.431 Da / 分子数: 2 / 断片: N terminal domain (residues 63-170) / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CEACAM1 / プラスミド: pGEX-2V / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CEACAM1 / プラスミド: pGEX-2V / 発現宿主:  #2: 化合物 | #3: 化合物 | #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.73 Å3/Da / 溶媒含有率: 54.95 % |
|---|---|
| 結晶化 | 温度: 294 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 詳細: 16% (w/v) polyethylene glycol mono methyl ether 2000, 100 mM Tris, and 5 mM nickel chloride, pH 8.5, VAPOR DIFFUSION, HANGING DROP, temperature 294K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 22-ID / 波長: 1 Å / ビームライン: 22-ID / 波長: 1 Å |
| 検出器 | タイプ: MARMOSAIC 300 mm CCD / 検出器: CCD / 日付: 2005年6月26日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.2→50 Å / Num. all: 13366 / Num. obs: 13366 / % possible obs: 99.6 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 10.5 % / Biso Wilson estimate: 50 Å2 / Rmerge(I) obs: 0.067 / Net I/σ(I): 38.8 |
| 反射 シェル | 解像度: 2.2→2.28 Å / 冗長度: 6.7 % / Rmerge(I) obs: 0.736 / Mean I/σ(I) obs: 2 / Num. unique all: 1290 / % possible all: 96.9 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1L6Z 解像度: 2.2→50 Å / Cor.coef. Fo:Fc: 0.941 / Cor.coef. Fo:Fc free: 0.909 / SU B: 11.806 / SU ML: 0.152 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.271 / ESU R Free: 0.218 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 41.5 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.2→2.257 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj










