+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2bj6 | ||||||
|---|---|---|---|---|---|---|---|
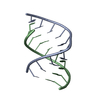
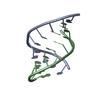
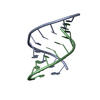
| タイトル | Crystal Structure of a decameric HNA-RNA hybrid | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | NUCLEIC ACID (HNA/RNA) / DNA / RNA / HNA / HEXITOL NUCLEIC ACID / HYBRID / DUPLEX / MODIFIED NUCLEIC ACID / ANTISENSE / BACKBONE MODIFICATION / NUCLEIC ACID (HNA-RNA) complex | ||||||
| 機能・相同性 | DNA / RNA 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.6 Å 多波長異常分散 / 解像度: 2.6 Å | ||||||
 データ登録者 データ登録者 | Maier, T. / Przylas, I. / Straeter, N. / Herdewijn, P. / Saenger, W. | ||||||
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2005 ジャーナル: J.Am.Chem.Soc. / 年: 2005タイトル: Reinforced Hna Backbone Hydration in the Crystal Structure of a Decameric Hna/RNA Hybrid 著者: Maier, T. / Przylas, I. / Strater, N. / Herdewijn, P. / Saenger, W. #1:  ジャーナル: J.Am.Chem.Soc. / 年: 1998 ジャーナル: J.Am.Chem.Soc. / 年: 1998タイトル: Molecular Dynamics Simulation to Inverstigate Differences in Minor Groove Hydration of Hna-RNA Hybrids as Compared to Hna-DNA Complexes 著者: Dewinter, H. / Lescrinier, E. / Vanaerschot, A. / Herdewijn, P. #2:  ジャーナル: Chem.Biol. / 年: 2000 ジャーナル: Chem.Biol. / 年: 2000タイトル: Solution Structure of a Hna-RNA Hybrid 著者: Lescrinier, E. / Esnouf, R. / Schraml, J. / Busson, R. / Heus, H.A. / Hilbers, C.W. / Herdewijn, P. #3:  ジャーナル: J.Am.Chem.Soc. / 年: 2002 ジャーナル: J.Am.Chem.Soc. / 年: 2002タイトル: Crystal Structure of Double Helical Hexitol Nucleic Acids 著者: Declercq, R. / Vanaerschot, A. / Read, R.J. / Herdewijn, P. / Vanmeervelt, L. #4:  ジャーナル: Acta Crystallogr.,Sect.C / 年: 1996 ジャーナル: Acta Crystallogr.,Sect.C / 年: 1996タイトル: 1,5-Anhydro-2,3-Dideoxy-2-(Guanin-9-Yl)-D-Arabino-Hexitol 著者: Declerq, R. / Herdewijn, P. / Vanmeervelt, L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2bj6.cif.gz 2bj6.cif.gz | 58.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2bj6.ent.gz pdb2bj6.ent.gz | 48.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2bj6.json.gz 2bj6.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2bj6_validation.pdf.gz 2bj6_validation.pdf.gz | 397.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2bj6_full_validation.pdf.gz 2bj6_full_validation.pdf.gz | 405.8 KB | 表示 | |
| XML形式データ |  2bj6_validation.xml.gz 2bj6_validation.xml.gz | 4.2 KB | 表示 | |
| CIF形式データ |  2bj6_validation.cif.gz 2bj6_validation.cif.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bj/2bj6 https://data.pdbj.org/pub/pdb/validation_reports/bj/2bj6 ftp://data.pdbj.org/pub/pdb/validation_reports/bj/2bj6 ftp://data.pdbj.org/pub/pdb/validation_reports/bj/2bj6 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 3145.249 Da / 分子数: 4 / 由来タイプ: 合成 / 詳細: CHEMICAL SYNTHESIS, PHOSPHOROTHIOATE / 由来: (合成)  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト)#2: RNA鎖 | 分子量: 3216.973 Da / 分子数: 4 / 由来タイプ: 合成 / 詳細: CHEMICAL SYNTHESIS, PHOSPHOROTHIOATE / 由来: (合成)  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト)#3: 化合物 | ChemComp-SO4 / #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.53 Å3/Da / 溶媒含有率: 66.2 % |
|---|---|
| 結晶化 | 詳細: 0.9M LISO4, 10MM MG(AC)2,0.1M TRIS/HCL, PH 8.0 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: BM30A / 波長: 0.92067 / ビームライン: BM30A / 波長: 0.92067 |
| 検出器 | タイプ: ADSC CCD / 検出器: CCD / 日付: 1999年11月25日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.92067 Å / 相対比: 1 |
| 反射 | 解像度: 2.54→35 Å / Num. obs: 11676 / % possible obs: 99.4 % / 冗長度: 6.64 % / Rmerge(I) obs: 0.06 / Net I/σ(I): 28.7 |
| 反射 シェル | 解像度: 2.54→2.63 Å / Mean I/σ(I) obs: 3.2 / % possible all: 95.5 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 2.6→35 Å / Data cutoff high absF: 10000 / 交差検証法: THROUGHOUT / σ(F): 0 多波長異常分散 / 解像度: 2.6→35 Å / Data cutoff high absF: 10000 / 交差検証法: THROUGHOUT / σ(F): 0 詳細: DIFFERENT ATOM NUMBERING SCHEMES ARE USED IN THE LITERATURE. PLEASE SEE PRIMARY AND SECONDARY CITATIONS FOR A DETAILED DESCRIPTION. STEREOCHEMICAL PARAMETERS FOR HNA MONOMERS HAVE BEEN ...詳細: DIFFERENT ATOM NUMBERING SCHEMES ARE USED IN THE LITERATURE. PLEASE SEE PRIMARY AND SECONDARY CITATIONS FOR A DETAILED DESCRIPTION. STEREOCHEMICAL PARAMETERS FOR HNA MONOMERS HAVE BEEN DERIVED FROM SMALL MOLECULE X-RAY STRUCTURES, SEE SECONDARY REFERENCES
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 48.1 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.6→35 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.6→2.64 Å / Total num. of bins used: 23 /
|
 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj




