+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2bhi | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of Taiwan cobra cardiotoxin A3 complexed with sulfogalactoceramide | ||||||
 要素 要素 | CYTOTOXIN 3 | ||||||
 キーワード キーワード | CARDIOTOXIN / COBRA CARDIOTOXIN / SULFOGALACTOCERAMIDE SULFATIDE / GLYCOSPHINGOLIPID / MEMBRANE PORE FORMATION / CYTOLYSIS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytolysis / other organism cell membrane / toxin activity / killing of cells of another organism / extracellular region / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  NAJA ATRA (タイワンコブラ) NAJA ATRA (タイワンコブラ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.31 Å 分子置換 / 解像度: 2.31 Å | ||||||
 データ登録者 データ登録者 | Wang, C.-H. / Liu, J.-H. / Wu, P.-L. / Lee, S.-C. / Hsiao, C.-D. / Wu, W.-G. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2006 ジャーナル: J.Biol.Chem. / 年: 2006タイトル: Glycosphingolipid-Facilitated Membrane Insertion and Internalization of Cobra Cardiotoxin: The Sulfatide/Cardiotoxin Complex Structure in a Membrane-Like Environment Suggests a Lipid- ...タイトル: Glycosphingolipid-Facilitated Membrane Insertion and Internalization of Cobra Cardiotoxin: The Sulfatide/Cardiotoxin Complex Structure in a Membrane-Like Environment Suggests a Lipid-Dependent Cell-Penetrating Mechanism for Membrane Binding Polypeptides. 著者: Wang, C.-H. / Liu, J.-H. / Lee, S.-C. / Hsiao, C.-D. / Wu, W.-G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2bhi.cif.gz 2bhi.cif.gz | 47.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2bhi.ent.gz pdb2bhi.ent.gz | 33.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2bhi.json.gz 2bhi.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2bhi_validation.pdf.gz 2bhi_validation.pdf.gz | 2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2bhi_full_validation.pdf.gz 2bhi_full_validation.pdf.gz | 2.1 MB | 表示 | |
| XML形式データ |  2bhi_validation.xml.gz 2bhi_validation.xml.gz | 10.8 KB | 表示 | |
| CIF形式データ |  2bhi_validation.cif.gz 2bhi_validation.cif.gz | 13.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bh/2bhi https://data.pdbj.org/pub/pdb/validation_reports/bh/2bhi ftp://data.pdbj.org/pub/pdb/validation_reports/bh/2bhi ftp://data.pdbj.org/pub/pdb/validation_reports/bh/2bhi | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1h0jS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 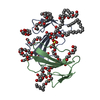
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6758.330 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  NAJA ATRA (タイワンコブラ) / 器官: VENOM GLAND / 参照: UniProt: P01444, UniProt: P60301*PLUS NAJA ATRA (タイワンコブラ) / 器官: VENOM GLAND / 参照: UniProt: P01444, UniProt: P60301*PLUS#2: 化合物 | ChemComp-SFT / | #3: 化合物 | ChemComp-C10 / #4: 水 | ChemComp-HOH / | 構成要素の詳細 | BELONGS TO THE SNAKE TOXIN FAMILY. | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.6 Å3/Da / 溶媒含有率: 52.2 % |
|---|---|
| 結晶化 | pH: 7 詳細: 1.8 M SODIUM MALONATE, 7.5% (V/V) TERT-BUTANOL, 7.5% (V/V) PENTAERYTHRITOL ETHOXYLATE (15/4 EO/OH), 0.08% (W/V) C10E6, 50 MM IMIDAZOLE, AND 50 MM TRIS-HCL, PH 7.0. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSRRC NSRRC  / ビームライン: BL17B2 / 波長: 1.1271 / ビームライン: BL17B2 / 波長: 1.1271 |
| 検出器 | タイプ: ADSC CCD / 検出器: CCD / 日付: 2004年4月18日 / 詳細: MIRRORS |
| 放射 | モノクロメーター: DCM WITH SAGITTAL FOCUSING / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.1271 Å / 相対比: 1 |
| 反射 | 解像度: 2.3→26.5 Å / Num. obs: 6821 / % possible obs: 99.8 % / Observed criterion σ(I): 2 / 冗長度: 6.1 % / Biso Wilson estimate: 25.9 Å2 / Rmerge(I) obs: 0.05 / Net I/σ(I): 34.6 |
| 反射 シェル | 解像度: 2.3→2.4 Å / 冗長度: 5.8 % / Rmerge(I) obs: 0.34 / Mean I/σ(I) obs: 5.1 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1H0J 解像度: 2.31→26.47 Å / Rfactor Rfree error: 0.01 / Data cutoff high absF: 521475.44 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 2 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 59.1255 Å2 / ksol: 0.370974 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 40.4 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.31→26.47 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.3→2.44 Å / Rfactor Rfree error: 0.025 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj