[English] 日本語
 Yorodumi
Yorodumi- PDB-1yct: Clustered abasic lesions in dna: nmr solution structure of cluste... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1yct | ||||||
|---|---|---|---|---|---|---|---|
| Title | Clustered abasic lesions in dna: nmr solution structure of clustered bistranded +1 abasic lesion | ||||||
 Components Components |
| ||||||
 Keywords Keywords | DNA / DOUBLE HELIX / ABASIC SITES / CLUSTERED DAMAGE / EXTRUDED RESIDUES / REDUCED AP SITE / FURAN / BISTRANDED ABASIC LESION. | ||||||
| Function / homology | DNA / DNA (> 10) Function and homology information Function and homology information | ||||||
| Method | SOLUTION NMR / RESTRAINED MOLECULAR DYNAMICS | ||||||
 Authors Authors | Hazel, R.D. / de los Santos, C. | ||||||
 Citation Citation |  Journal: Biochemistry / Year: 2008 Journal: Biochemistry / Year: 2008Title: NMR solution structures of bistranded abasic site lesions in DNA. Authors: Hazel, R.D. / Tian, K. / de Los Santos, C. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1yct.cif.gz 1yct.cif.gz | 89.1 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1yct.ent.gz pdb1yct.ent.gz | 69.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1yct.json.gz 1yct.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/yc/1yct https://data.pdbj.org/pub/pdb/validation_reports/yc/1yct ftp://data.pdbj.org/pub/pdb/validation_reports/yc/1yct ftp://data.pdbj.org/pub/pdb/validation_reports/yc/1yct | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 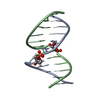
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR ensembles |
|
- Components
Components
| #1: DNA chain | Mass: 3843.488 Da / Num. of mol.: 1 / Source method: obtained synthetically Details: RANDOMLY DESIGNED SEQUENCE CONTAINING STABLE ABASIC SITES (3DR) |
|---|---|
| #2: DNA chain | Mass: 3843.488 Da / Num. of mol.: 1 / Source method: obtained synthetically Details: RANDOMLY DESIGNED SEQUENCE CONTAINING STABLE ABASIC SITES (3DR) |
-Experimental details
-Experiment
| Experiment | Method: SOLUTION NMR | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR experiment |
| ||||||||||||||||||||
| NMR details | Text: This structure was determined using standard 2D homonuclear techniques. |
- Sample preparation
Sample preparation
| Details | Contents: 2mM DUPLEX, 25 mM PHOSPHATE BUFFER, 50 mM NaCl, 0.5 mM EDTA. Solvent system: 25 mM PHOSPHATE BUFFER, PH 6.8; 50 mM NaCl; 0.5 mM EDTA. |
|---|---|
| Sample conditions | Ionic strength: NOT DETERMINED / pH: 6.8 / Pressure: AMBIENT / Temperature: 298 K |
-NMR measurement
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Radiation wavelength | Relative weight: 1 | |||||||||||||||
| NMR spectrometer |
|
- Processing
Processing
| NMR software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method: RESTRAINED MOLECULAR DYNAMICS / Software ordinal: 1 Details: 1) HEATING STAGE: FROM 150 TO 500 K IN 70PS. INTRODUCTION OF EXPERIMENTAL RESTRAINTS AT THE END OF THIS STAGE. 2) HIGH TEMPERATURE STAGE: 50 PS DYNAMICS AT 500K 3) COOLING STAGE: FROM 500 TO ...Details: 1) HEATING STAGE: FROM 150 TO 500 K IN 70PS. INTRODUCTION OF EXPERIMENTAL RESTRAINTS AT THE END OF THIS STAGE. 2) HIGH TEMPERATURE STAGE: 50 PS DYNAMICS AT 500K 3) COOLING STAGE: FROM 500 TO 300K IN 40PS. 4) EQUILIBRATION STAGE: 120PS DYNAMICS AT 300K | ||||||||||||||||||||||||
| NMR representative | Selection criteria: closest to the average | ||||||||||||||||||||||||
| NMR ensemble | Conformer selection criteria: structures with the least restraint violations Conformers calculated total number: 20 / Conformers submitted total number: 5 |
 Movie
Movie Controller
Controller









 PDBj
PDBj






































 X-PLOR
X-PLOR