+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1s75 | ||||||
|---|---|---|---|---|---|---|---|
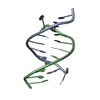
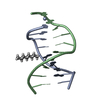
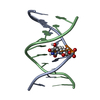
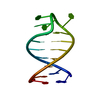
| タイトル | SOLUTION STRUCTURE OF A DNA DUPLEX CONTAINING AN ALPHA-ANOMERIC ADENOSINE: INSIGHTS INTO SUBSTRATE RECOGNITION BY ENDONUCLEASE IV | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA / DNA DOUBLE HELIX WITH ENLARGED MINER GROOVE AND HELICAL KINK | ||||||
| 機能・相同性 | DNA 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / TORSION ANGLE DYNAMICS, MOLECULAR DYNAMICS | ||||||
| Model type details | minimized average | ||||||
 データ登録者 データ登録者 | Aramini, J.M. / Cleaver, S.H. / Pon, R.T. / Cunningham, R.P. / Germann, M.W. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2004 ジャーナル: J.Mol.Biol. / 年: 2004タイトル: Solution Structure of a DNA Duplex Containing an alpha-Anomeric Adenosine: Insights into Substrate Recognition by Endonuclease IV. 著者: Aramini, J.M. / Cleaver, S.H. / Pon, R.T. / Cunningham, R.P. / Germann, M.W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1s75.cif.gz 1s75.cif.gz | 22.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1s75.ent.gz pdb1s75.ent.gz | 14.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1s75.json.gz 1s75.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1s75_validation.pdf.gz 1s75_validation.pdf.gz | 252.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1s75_full_validation.pdf.gz 1s75_full_validation.pdf.gz | 251.9 KB | 表示 | |
| XML形式データ |  1s75_validation.xml.gz 1s75_validation.xml.gz | 1.9 KB | 表示 | |
| CIF形式データ |  1s75_validation.cif.gz 1s75_validation.cif.gz | 2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/s7/1s75 https://data.pdbj.org/pub/pdb/validation_reports/s7/1s75 ftp://data.pdbj.org/pub/pdb/validation_reports/s7/1s75 ftp://data.pdbj.org/pub/pdb/validation_reports/s7/1s75 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 3029.994 Da / 分子数: 1 / 由来タイプ: 合成 詳細: THE CORE OF THE SEQUENCE CORRESPONDS TO THE RECOGNITION SITE OF E. COLI ENDONUCLEASE IV |
|---|---|
| #2: DNA鎖 | 分子量: 3061.003 Da / 分子数: 1 / 由来タイプ: 合成 |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: FOR NON-EXCHANGEABLE PROTONS, NOESY EXPERIMENTS IN D2O WERE PERFORMED WITH 10S RELAXATION DELAYS AND MIXING TIMES OF 75MS, 150MS AND 250 MS. FOR EXCHANGEABLE PROTONS, A WATERGATE NOESY WAS ...Text: FOR NON-EXCHANGEABLE PROTONS, NOESY EXPERIMENTS IN D2O WERE PERFORMED WITH 10S RELAXATION DELAYS AND MIXING TIMES OF 75MS, 150MS AND 250 MS. FOR EXCHANGEABLE PROTONS, A WATERGATE NOESY WAS PERFORMED WITH A RELAXATION DELAY OF 4S AND 150MS MIXING TIME. |
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
|
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射波長 | 相対比: 1 | |||||||||||||||
| NMRスペクトロメーター |
|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: TORSION ANGLE DYNAMICS, MOLECULAR DYNAMICS / ソフトェア番号: 1 詳細: THE STRUCTURE IS BASED ON A TOTAL OF 502 RESTRAINTS: 284 DISTANCE RESTRAINTS, 80 ENDOCYCLIC TORSION ANGLE RESTRAINTS, 50 WATSON-CRICK DISTANCE AND ANGLE RESTRAINTS, AND 88 BACKBONE TORSION ...詳細: THE STRUCTURE IS BASED ON A TOTAL OF 502 RESTRAINTS: 284 DISTANCE RESTRAINTS, 80 ENDOCYCLIC TORSION ANGLE RESTRAINTS, 50 WATSON-CRICK DISTANCE AND ANGLE RESTRAINTS, AND 88 BACKBONE TORSION ANGLE RESTRAINTS. THE ALPHAA DUPLEX STRUCTURE WAS ELUCIDATED BY A COMBINATION OF DYANA AND RMD/REM IN AMBER. ALL CALCULATIONS WERE PERFORMED IN VACUO. THE FINAL STRUCTURE DEPOSITED HERE WAS OBTAINED BY COORDINATE AVERAGING THE FINAL ENSEMBLE OF 10 RMD/REM STRUCTURES FOLLOWED BY RESTRAINED ENERGY MINIMIZATION. THE IN VACUO STRUCTURE WAS THEN SOLVATED IN A SOLVENT BOX AND NA+ COUNTERIONS. THE FOLLOWING SIMULATIONS CONTAINED 25 C-H RESIDUAL DIPOLAR COUPLINGS RESTRAINTS. THE FINAL SOLVATED STRUCTURE WAS OBTAINED BY AVERAGING THE LAST 5PS OF A 1 NS RMD RUN FOLLOWED BY REM. | ||||||||||||||||||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||||||||||||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj

 HSQC
HSQC