+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1q8n | ||||||
|---|---|---|---|---|---|---|---|
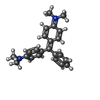
| Title | Solution Structure of the Malachite Green RNA Binding Aptamer | ||||||
 Components Components | RNA Aptamer | ||||||
 Keywords Keywords | RNA / rna aptamer / malachite green / base triple / base quadruple / rna ligand interactions | ||||||
| Function / homology | MALACHITE GREEN / RNA / RNA (> 10) Function and homology information Function and homology information | ||||||
| Method | SOLUTION NMR / simulated annealing | ||||||
 Authors Authors | Flinders, J. / DeFina, S.C. / Brackett, D.M. / Baugh, C. / Wilson, C. / Dieckmann, T. | ||||||
 Citation Citation |  Journal: Chembiochem / Year: 2004 Journal: Chembiochem / Year: 2004Title: Recognition of planar and nonplanar ligands in the malachite green-RNA aptamer complex. Authors: Flinders, J. / DeFina, S.C. / Brackett, D.M. / Baugh, C. / Wilson, C. / Dieckmann, T. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1q8n.cif.gz 1q8n.cif.gz | 606.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1q8n.ent.gz pdb1q8n.ent.gz | 509.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1q8n.json.gz 1q8n.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/q8/1q8n https://data.pdbj.org/pub/pdb/validation_reports/q8/1q8n ftp://data.pdbj.org/pub/pdb/validation_reports/q8/1q8n ftp://data.pdbj.org/pub/pdb/validation_reports/q8/1q8n | HTTPS FTP |
|---|
-Related structure data
| Related structure data | |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR ensembles |
|
- Components
Components
| #1: RNA chain | Mass: 12317.421 Da / Num. of mol.: 1 / Source method: obtained synthetically Details: This sequence was synthesized in vitro and does not occur naturally. |
|---|---|
| #2: Chemical | ChemComp-MGR / |
-Experimental details
-Experiment
| Experiment | Method: SOLUTION NMR | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR experiment |
| ||||||||||||||||||||||||||||||||
| NMR details | Text: This structure was determined with the aid of the crystal structure of a similar RNA aptamer. |
- Sample preparation
Sample preparation
| Details |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Sample conditions |
| ||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Method: other / Details: NMR |
-NMR measurement
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M |
|---|---|
| Radiation wavelength | Relative weight: 1 |
| NMR spectrometer | Type: Bruker DRX / Manufacturer: Bruker / Model: DRX / Field strength: 600 MHz |
- Processing
Processing
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method: simulated annealing / Software ordinal: 1 Details: The structure was based on 423 NOE constraints (148 intranucleotide, 119 sequential, 66 medium to long range, and 90 RNA to ligand NOEs) and 219 dihedral angle restraints. | ||||||||||||||||||||
| NMR representative | Selection criteria: lowest energy | ||||||||||||||||||||
| NMR ensemble | Conformer selection criteria: structures with the lowest energy Conformers calculated total number: 100 / Conformers submitted total number: 25 |
 Movie
Movie Controller
Controller












 PDBj
PDBj