+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ngo | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | NMR Structure of Putative 3' Terminator for B. Anthracis pagA Gene Coding Strand | ||||||||||||||||||||
 要素 要素 | 5'-D(* キーワード キーワードDNA / B-Form DNA Hairpin | 機能・相同性 | DNA / DNA (> 10) |  機能・相同性情報 機能・相同性情報手法 | 溶液NMR / minimization molecular dynamics | Model type details | minimized average |  データ登録者 データ登録者Shiflett, P.R. / Taylor-McCabe, K.J. / Michalczyk, R. / Silks, L.A. / Gupta, G. |  引用 引用 ジャーナル: Biochemistry / 年: 2003 ジャーナル: Biochemistry / 年: 2003タイトル: Structural Studies on the Hairpins at the 3' Untranslated Region of an Anthrax Toxin Gene 著者: Shiflett, P.R. / Taylor-McCabe, K.J. / Michalczyk, R. / Silks, L.A. / Gupta, G. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ngo.cif.gz 1ngo.cif.gz | 75.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ngo.ent.gz pdb1ngo.ent.gz | 60.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ngo.json.gz 1ngo.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1ngo_validation.pdf.gz 1ngo_validation.pdf.gz | 310 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1ngo_full_validation.pdf.gz 1ngo_full_validation.pdf.gz | 320.2 KB | 表示 | |
| XML形式データ |  1ngo_validation.xml.gz 1ngo_validation.xml.gz | 2.4 KB | 表示 | |
| CIF形式データ |  1ngo_validation.cif.gz 1ngo_validation.cif.gz | 3.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ng/1ngo https://data.pdbj.org/pub/pdb/validation_reports/ng/1ngo ftp://data.pdbj.org/pub/pdb/validation_reports/ng/1ngo ftp://data.pdbj.org/pub/pdb/validation_reports/ng/1ngo | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 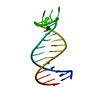
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 8363.434 Da / 分子数: 1 / 由来タイプ: 合成 詳細: This sequence occurs on the coding strand of the 3' UTR of the pagA (110) gene of Bacillus anthracis. |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||
| NMR実験の詳細 | Text: The structure was determined using standard 2D homonuclear techniques. |
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
| |||||||||||||||
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Bruker AVANCE / 製造業者: Bruker / モデル: AVANCE / 磁場強度: 500 MHz |
- 解析
解析
| NMR software |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: minimization molecular dynamics / ソフトェア番号: 1 詳細: Structure is derived from 191 NOES of which, 25 involve exchangeable protons in the stem, 153 NOEs involve non-exchangeable protons in the stem, 13 NOEs involve non-exchangeable protons in ...詳細: Structure is derived from 191 NOES of which, 25 involve exchangeable protons in the stem, 153 NOEs involve non-exchangeable protons in the stem, 13 NOEs involve non-exchangeable protons in the loop, 50 Distance restraints involve hydrogen bonds, and 44 dihedral angle restraints. | ||||||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: Submitted conformers include average from molecular dynamics and 3 representative diverging structures. 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 4 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj






































 Amber
Amber