+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1mfj | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
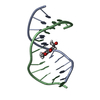
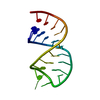
| Title | 3' Stem-Loop from Human U4 SNRNA | ||||||||||||||||||
 Components Components | 5'-R(* Keywords KeywordsRNA / RIBONUCLEIC ACID / RNA OLIGONUCLEOTIDE / STEM-AND-LOOP / U4 SMALL NUCLEAR RNA / UACG TETRALOOP | Function / homology | RNA / RNA (> 10) |  Function and homology information Function and homology informationMethod | SOLUTION NMR / complete relaxation matrix; random error analysis of NOE; torsion angle dynamics; simulated annealing using Metropolis Monte Carlo; restrained minimization |  Authors AuthorsComolli, L.R. / Ulyanov, N.B. / James, T.L. / Gmeiner, W.H. |  Citation Citation Journal: Nucleic Acids Res. / Year: 2002 Journal: Nucleic Acids Res. / Year: 2002Title: NMR Structure of the 3' Stem-Loop from Human U4 snRNA Authors: Comolli, L.R. / Ulyanov, N.B. / Soto, A.M. / Marky, L.A. / James, T.L. / Gmeiner, W.H. History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1mfj.cif.gz 1mfj.cif.gz | 127.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1mfj.ent.gz pdb1mfj.ent.gz | 106.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1mfj.json.gz 1mfj.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/mf/1mfj https://data.pdbj.org/pub/pdb/validation_reports/mf/1mfj ftp://data.pdbj.org/pub/pdb/validation_reports/mf/1mfj ftp://data.pdbj.org/pub/pdb/validation_reports/mf/1mfj | HTTPS FTP |
|---|
-Related structure data
| Similar structure data |
|---|
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR ensembles |
|
- Components
Components
| #1: RNA chain | Mass: 6422.879 Da / Num. of mol.: 1 / Source method: obtained synthetically / Details: This sequence is part of human U4 snRNA. |
|---|
-Experimental details
-Experiment
| Experiment | Method: SOLUTION NMR | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR experiment |
| ||||||||||||||||||||||||
| NMR details | Text: This structure was determined using standard 2D homonuclear techniques and complete relaxation matrix analysis of NOE intensities. |
- Sample preparation
Sample preparation
| Details |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Sample conditions |
| |||||||||||||||
| Crystal grow | *PLUS Method: other / Details: NMR |
-NMR measurement
| NMR spectrometer | Type: Varian INOVA / Manufacturer: Varian / Model: INOVA / Field strength: 600 MHz |
|---|
- Processing
Processing
| NMR software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method: complete relaxation matrix; random error analysis of NOE; torsion angle dynamics; simulated annealing using Metropolis Monte Carlo; restrained minimization Software ordinal: 1 Details: Structures are based on 200 distance restraints, of which 140 are quantitative bounds for nonexchangeable protons calculated with MARDIGRAS, 56 are upper bounds for exchangeable protons, and ...Details: Structures are based on 200 distance restraints, of which 140 are quantitative bounds for nonexchangeable protons calculated with MARDIGRAS, 56 are upper bounds for exchangeable protons, and 4 are hydrogen bond restraints. | ||||||||||||||||||||||||||||
| NMR representative | Selection criteria: lowest total energy | ||||||||||||||||||||||||||||
| NMR ensemble | Conformer selection criteria: Lowest total energy (a weighted sum of conformational energy and restraint energy). Conformers calculated total number: 15 / Conformers submitted total number: 10 |
 Movie
Movie Controller
Controller





 PDBj
PDBj





























 NMRPipe
NMRPipe