+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1kve | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | KILLER TOXIN FROM HALOTOLERANT YEAST | ||||||
 要素 要素 | (SMK TOXIN) x 2 | ||||||
 キーワード キーワード | TOXIN / HALOTOLERANT YEAST | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報toxin sequestering activity / toxin activity / killing of cells of another organism / protein-containing complex / extracellular region 類似検索 - 分子機能 | ||||||
| 生物種 |  Pichia farinosa (菌類) Pichia farinosa (菌類) | ||||||
| 手法 |  X線回折 / 解像度: 1.8 Å X線回折 / 解像度: 1.8 Å | ||||||
 データ登録者 データ登録者 | Kashiwagi, T. / Kunishima, N. / Suzuki, C. / Tsuchiya, F. / Nikkuni, S. / Arata, Y. / Morikawa, K. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: The novel acidophilic structure of the killer toxin from halotolerant yeast demonstrates remarkable folding similarity with a fungal killer toxin. 著者: Kashiwagi, T. / Kunishima, N. / Suzuki, C. / Tsuchiya, F. / Nikkuni, S. / Arata, Y. / Morikawa, K. #1:  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Crystallization and Preliminary X-Ray Diffraction Studies of a Novel Killer Toxin from a Halotolerant Yeast Pichia Farinosa 著者: Kunishima, N. / Kashiwagi, T. / Suzuki, C. / Nikkuni, S. / Tsuchiya, F. / Arata, Y. / Morikawa, K. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1kve.cif.gz 1kve.cif.gz | 66.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1kve.ent.gz pdb1kve.ent.gz | 50.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1kve.json.gz 1kve.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1kve_validation.pdf.gz 1kve_validation.pdf.gz | 427.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1kve_full_validation.pdf.gz 1kve_full_validation.pdf.gz | 427.6 KB | 表示 | |
| XML形式データ |  1kve_validation.xml.gz 1kve_validation.xml.gz | 17.5 KB | 表示 | |
| CIF形式データ |  1kve_validation.cif.gz 1kve_validation.cif.gz | 24 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kv/1kve https://data.pdbj.org/pub/pdb/validation_reports/kv/1kve ftp://data.pdbj.org/pub/pdb/validation_reports/kv/1kve ftp://data.pdbj.org/pub/pdb/validation_reports/kv/1kve | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 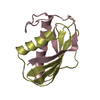
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
| ||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (0.09656, -0.87326, 0.47759), ベクター: |
- 要素
要素
| #1: タンパク質 | 分子量: 6349.229 Da / 分子数: 2 断片: CHAIN A AND C ARE RESIDUES 19 - 81 OF THE ALPHA CHAIN, CHAIN B AND D ARE RESIDUES 146 - 222 OF THE BETA CHAIN 由来タイプ: 天然 / 由来: (天然)  Pichia farinosa (菌類) / 株: KK1 / 参照: UniProt: P19972 Pichia farinosa (菌類) / 株: KK1 / 参照: UniProt: P19972#2: タンパク質 | 分子量: 7856.556 Da / 分子数: 2 断片: CHAIN A AND C ARE RESIDUES 19 - 81 OF THE ALPHA CHAIN, CHAIN B AND D ARE RESIDUES 146 - 222 OF THE BETA CHAIN 由来タイプ: 天然 / 由来: (天然)  Pichia farinosa (菌類) / 株: KK1 / 参照: UniProt: P19972 Pichia farinosa (菌類) / 株: KK1 / 参照: UniProt: P19972#3: 水 | ChemComp-HOH / | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.43 Å3/Da / 溶媒含有率: 64.2 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 4 詳細: CRYSTALS WERE GROWN FROM 25% POLYETHYLENE GLYCOL SOLUTION AT PH 4.0. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射光源 | 波長: 1.5418 |
|---|---|
| 検出器 | タイプ: MACSCIENCE / 検出器: IMAGE PLATE / 日付: 1995年11月20日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→50 Å / Num. obs: 35468 / % possible obs: 94.7 % / Observed criterion σ(I): 0.5 / 冗長度: 5.23 % / Rmerge(I) obs: 0.062 |
| 反射 | *PLUS Num. measured all: 185448 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.8→6 Å / σ(F): 1 詳細: IDEAL BOND LENGTHS AND ANGLES USED DURING REFINEMENT: HENDRICKSON AND KONNERT FINAL RMS COORD. SHIFT 0.007 ANGSTROMS INITIAL REFINEMENTS WERE DONE WITH X-PLOR 3.1 BY BRUNGER.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 22.49 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→6 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: PROLSQ / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.172 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
