+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1byj | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
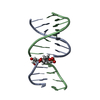
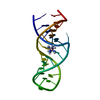
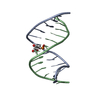
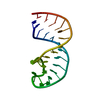
| タイトル | GENTAMICIN C1A A-SITE COMPLEX | |||||||||
 要素 要素 | RNA (16S RNA) | |||||||||
 キーワード キーワード | RNA / COMPLEX (AMINOGLYCOSIDE-RIBOSOMAL RNA) | |||||||||
| 機能・相同性 | Chem-GE1 / 3,5-DIAMINO-CYCLOHEXANOL / Chem-GE3 / RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | |||||||||
| 手法 | 溶液NMR / simulated annealing | |||||||||
 データ登録者 データ登録者 | Yoshizawa, S. / Fourmy, D. / Puglisi, J.D. | |||||||||
 引用 引用 |  ジャーナル: EMBO J. / 年: 1998 ジャーナル: EMBO J. / 年: 1998タイトル: Structural origins of gentamicin antibiotic action. 著者: Yoshizawa, S. / Fourmy, D. / Puglisi, J.D. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1byj.cif.gz 1byj.cif.gz | 788.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1byj.ent.gz pdb1byj.ent.gz | 575.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1byj.json.gz 1byj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1byj_validation.pdf.gz 1byj_validation.pdf.gz | 379.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1byj_full_validation.pdf.gz 1byj_full_validation.pdf.gz | 544.2 KB | 表示 | |
| XML形式データ |  1byj_validation.xml.gz 1byj_validation.xml.gz | 115.4 KB | 表示 | |
| CIF形式データ |  1byj_validation.cif.gz 1byj_validation.cif.gz | 141.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/by/1byj https://data.pdbj.org/pub/pdb/validation_reports/by/1byj ftp://data.pdbj.org/pub/pdb/validation_reports/by/1byj ftp://data.pdbj.org/pub/pdb/validation_reports/by/1byj | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 8656.185 Da / 分子数: 1 / 断片: DECODING REGION / 由来タイプ: 合成 / 詳細: 16S RNA WAS CHEMICALLY SYNTHESIZED. |
|---|---|
| #2: 糖 | ChemComp-GE1 / |
| #3: 化合物 | ChemComp-GE2 / |
| #4: 糖 | ChemComp-GE3 / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||
| NMR実験の詳細 | Text: A TOTAL OF 326 RNA, 7 GENTAMICIN AND 46 GENTAMICIN-RNA RESTRAINTS WERE USED. 111 EXPERIMENTAL DIHEDRAL RESTRAINTS WERE USED. FOR 38 THE FINAL STRUCTURES DEVIATIONS FROM EXPERIMENTAL RESTRAINTS ...Text: A TOTAL OF 326 RNA, 7 GENTAMICIN AND 46 GENTAMICIN-RNA RESTRAINTS WERE USED. 111 EXPERIMENTAL DIHEDRAL RESTRAINTS WERE USED. FOR 38 THE FINAL STRUCTURES DEVIATIONS FROM EXPERIMENTAL RESTRAINTS ARE: RMSD NMR DISTANCE RESTRAINTS (379) (A): 0.0249 (0.0002) RMSD NMR DIHEDRAL RESTRAINTS (111) (DEG): 0.0127 (0.0016) DEVIATIONS FROM DEALIZED GEOMETRY (CVFF FORCEFIELD) RMSD BONDS (A): 0.0260 ( 0.0000) RMSD ANGLES (DEG): 0.0621 (0.0001) RMSD IMPROPERS (DEG): 0.0485 ( 0.0025) HEAVY ATOM RMS DEVIATIONS OF AVERAGE, MINIMIZED STRUCTURE FROM BEST- FIT SUPERPOSITION OF THE 38 FINAL STRUCTURES RMSD ALL RNA + GENTAMICIN (A): 0.89 |
- 試料調製
試料調製
| 試料状態 | イオン強度: 10 mM SODIUM PHOSPHATE / pH: 6.4 / 温度: 308 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION 計算したコンフォーマーの数: 38 / 登録したコンフォーマーの数: 38 |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj