+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9378 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PGT145 Fab in complex with full length AMC011 HIV-1 Env | ||||||||||||||||||
 マップデータ マップデータ | PGT145 Fab in complex with full length AMC011 HIV-1 Env | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | HIV-1 / antibody / glycoprotein / IMMUNE SYSTEM | ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.7 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Torrents de la Pena A / Rantalainen K / Torres JL / Ward AB | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2019 ジャーナル: PLoS Pathog / 年: 2019タイトル: Similarities and differences between native HIV-1 envelope glycoprotein trimers and stabilized soluble trimer mimetics. 著者: Alba Torrents de la Peña / Kimmo Rantalainen / Christopher A Cottrell / Joel D Allen / Marit J van Gils / Jonathan L Torres / Max Crispin / Rogier W Sanders / Andrew B Ward /    要旨: The HIV-1 envelope glycoprotein (Env) trimer is located on the surface of the virus and is the target of broadly neutralizing antibodies (bNAbs). Recombinant native-like soluble Env trimer mimetics, ...The HIV-1 envelope glycoprotein (Env) trimer is located on the surface of the virus and is the target of broadly neutralizing antibodies (bNAbs). Recombinant native-like soluble Env trimer mimetics, such as SOSIP trimers, have taken a central role in HIV-1 vaccine research aimed at inducing bNAbs. We therefore performed a direct and thorough comparison of a full-length unmodified Env trimer containing the transmembrane domain and the cytoplasmic tail, with the sequence matched soluble SOSIP trimer, both based on an early Env sequence (AMC011) from an HIV+ individual that developed bNAbs. The structures of the full-length AMC011 trimer bound to either bNAb PGT145 or PGT151 were very similar to the structures of SOSIP trimers. Antigenically, the full-length and SOSIP trimers were comparable, but in contrast to the full-length trimer, the SOSIP trimer did not bind at all to non-neutralizing antibodies, most likely as a consequence of the intrinsic stabilization of the SOSIP trimer. Furthermore, the glycan composition of full-length and SOSIP trimers was similar overall, but the SOSIP trimer possessed slightly less complex and less extensively processed glycans, which may relate to the intrinsic stabilization as well as the absence of the membrane tether. These data provide insights into how to best use and improve membrane-associated full-length and soluble SOSIP HIV-1 Env trimers as immunogens. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9378.map.gz emd_9378.map.gz | 117.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9378-v30.xml emd-9378-v30.xml emd-9378.xml emd-9378.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9378.png emd_9378.png | 31.8 KB | ||
| Filedesc metadata |  emd-9378.cif.gz emd-9378.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9378 http://ftp.pdbj.org/pub/emdb/structures/EMD-9378 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9378 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9378 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9378_validation.pdf.gz emd_9378_validation.pdf.gz | 493.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9378_full_validation.pdf.gz emd_9378_full_validation.pdf.gz | 492.7 KB | 表示 | |
| XML形式データ |  emd_9378_validation.xml.gz emd_9378_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  emd_9378_validation.cif.gz emd_9378_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9378 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9378 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9378 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9378 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9378.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9378.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PGT145 Fab in complex with full length AMC011 HIV-1 Env | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
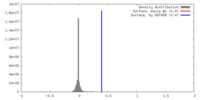
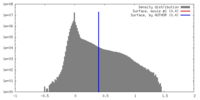
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PGT145 Fab in complex with full length AMC011 HIV-1 Env
| 全体 | 名称: PGT145 Fab in complex with full length AMC011 HIV-1 Env |
|---|---|
| 要素 |
|
-超分子 #1: PGT145 Fab in complex with full length AMC011 HIV-1 Env
| 超分子 | 名称: PGT145 Fab in complex with full length AMC011 HIV-1 Env タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: PGT145
| 超分子 | 名称: PGT145 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: AMC011 Glycoprotein 120
| 超分子 | 名称: AMC011 Glycoprotein 120 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #4: AMC011 Glycoprotein 41
| 超分子 | 名称: AMC011 Glycoprotein 41 / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: PGT145 Fab heavy chain
| 分子 | 名称: PGT145 Fab heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 15.550196 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGAE VKKPGSSVKV SCKASGNSFS NHDVHWVRQA TGQGLEWMGW MSHEGDKTGL AQKFQGRVTI TRDSGASTVY MELRGLTAD DTAIYYCLTG SKHRLRDYFL YNEYGPNYEE WGDYLATLDV WGHGTAVTVS S |
-分子 #2: PGT145 Fab light chain
| 分子 | 名称: PGT145 Fab light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.350922 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVVITQSPLF LPVTPGEAAS LSCKCSHSLQ HSTGANYLAW YLQRPGQTPR LLIHLATHRA SGVPDRFSGS GSGTDFTLKI SRVESDDVG TYYCMQGLHS PWTFGQGTKV EIKR |
-分子 #3: AMC011 Glycoprotein 120
| 分子 | 名称: AMC011 Glycoprotein 120 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 52.880039 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AEQLWVTVYY GVPVWKEATT TLFCASDARA YDTEVHNVWA THACVPTDPN PQEVVLENVT ENFNMWKNNM VEQMHEDIIS LWDQSLKPC VKLTPLCVTL NCTDLRNATN TNATNTTSSS RGTMEGGEIK NCSFNITTSM RDKVQKEYAL FYKLDVVPIK N DNTSYRLI ...文字列: AEQLWVTVYY GVPVWKEATT TLFCASDARA YDTEVHNVWA THACVPTDPN PQEVVLENVT ENFNMWKNNM VEQMHEDIIS LWDQSLKPC VKLTPLCVTL NCTDLRNATN TNATNTTSSS RGTMEGGEIK NCSFNITTSM RDKVQKEYAL FYKLDVVPIK N DNTSYRLI SCNTSVITQA CPKVSFEPIP IHYCAPAGFA ILKCNDKKFN GTGPCTNVST VQCTHGIRPV VSTQLLLNGS LA EEEVVIR SANFTDNAKI IIVQLNKSVE INCTRPNNNT RKSIHIGPGR AFYTTGEIIG DIRQAHCNIS GTKWNDTLKQ IVV KLKEQF GNKTIVFNHS SGGDPEIVMH SFNCGGEFFY CNSTQLFNST WNDTTGSNYT GTIVLPCRIK QIVNMWQEVG KAMY APPIK GQIRCSSNIT GLILIRDGGK NRSENTEIFR PGGGDMRDNW RSELYKYKVV KIEPLGIAPT KAKRRVVQ |
-分子 #4: AMC011 Glycoprotein 41
| 分子 | 名称: AMC011 Glycoprotein 41 / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 39.677762 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RLLLSGIVQQ QNNLLRAIEA QQHLLQLTVW GIKQLQARVL AVERYLKDQQ LLGIWGCSG KLICTTAVPW NTSWSNKSYN QIWNNMTWME WEREIDNYTS LIYTLIEDSQ NQQEKNEQEL LELDKWASLW N WFDITKWL ...文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RLLLSGIVQQ QNNLLRAIEA QQHLLQLTVW GIKQLQARVL AVERYLKDQQ LLGIWGCSG KLICTTAVPW NTSWSNKSYN QIWNNMTWME WEREIDNYTS LIYTLIEDSQ NQQEKNEQEL LELDKWASLW N WFDITKWL WYIKIFIMIV GGLIGLRIVF TVLSIVNRIR QGYSPLSFQT PLPTPRGPDR PEGIEEEGGE RDRDRSDRLV TG FLALIWV DLRSLCLFSY HRLRDLLLIV TRIVELLGRR GWGVLKYWWN LLQYWSQELR NSAVSLLNAT AIAVAEGTDR VIE VSQRAF RAILHVPVRI RQGLERALV |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 5 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 5 sec. |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 5.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 51588 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6nij: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)