+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9030 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV-1 Envelope Glycoprotein Clone B41_delCT in complex with the broadly neutralizing antibody Fab PGT151 | |||||||||
 マップデータ マップデータ | primary map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
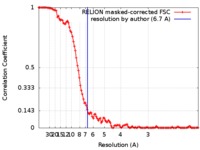
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.7 Å | |||||||||
 データ登録者 データ登録者 | Berndsen ZT / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: Differential processing of HIV envelope glycans on the virus and soluble recombinant trimer. 著者: Liwei Cao / Matthias Pauthner / Raiees Andrabi / Kimmo Rantalainen / Zachary Berndsen / Jolene K Diedrich / Sergey Menis / Devin Sok / Raiza Bastidas / Sung-Kyu Robin Park / Claire M ...著者: Liwei Cao / Matthias Pauthner / Raiees Andrabi / Kimmo Rantalainen / Zachary Berndsen / Jolene K Diedrich / Sergey Menis / Devin Sok / Raiza Bastidas / Sung-Kyu Robin Park / Claire M Delahunty / Lin He / Javier Guenaga / Richard T Wyatt / William R Schief / Andrew B Ward / John R Yates / Dennis R Burton / James C Paulson /  要旨: As the sole target of broadly neutralizing antibodies (bnAbs) to HIV, the envelope glycoprotein (Env) trimer is the focus of vaccination strategies designed to elicit protective bnAbs in humans. ...As the sole target of broadly neutralizing antibodies (bnAbs) to HIV, the envelope glycoprotein (Env) trimer is the focus of vaccination strategies designed to elicit protective bnAbs in humans. Because HIV Env is densely glycosylated with 75-90 N-glycans per trimer, most bnAbs use or accommodate them in their binding epitope, making the glycosylation of recombinant Env a key aspect of HIV vaccine design. Upon analysis of three HIV strains, we here find that site-specific glycosylation of Env from infectious virus closely matches Envs from corresponding recombinant membrane-bound trimers. However, viral Envs differ significantly from recombinant soluble, cleaved (SOSIP) Env trimers, strongly impacting antigenicity. These results provide a benchmark for virus Env glycosylation needed for the design of soluble Env trimers as part of an overall HIV vaccine strategy. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9030.map.gz emd_9030.map.gz | 116.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9030-v30.xml emd-9030-v30.xml emd-9030.xml emd-9030.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9030_fsc.xml emd_9030_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9030.png emd_9030.png | 59.2 KB | ||
| その他 |  emd_9030_half_map_1.map.gz emd_9030_half_map_1.map.gz emd_9030_half_map_2.map.gz emd_9030_half_map_2.map.gz | 98.4 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9030 http://ftp.pdbj.org/pub/emdb/structures/EMD-9030 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9030 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9030 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9030_validation.pdf.gz emd_9030_validation.pdf.gz | 79 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9030_full_validation.pdf.gz emd_9030_full_validation.pdf.gz | 78.1 KB | 表示 | |
| XML形式データ |  emd_9030_validation.xml.gz emd_9030_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9030 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9030 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9030 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9030 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9030.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9030.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
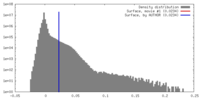
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 1
| ファイル | emd_9030_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
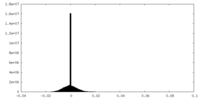
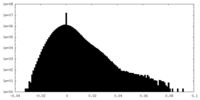
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_9030_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
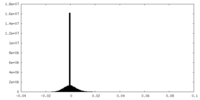
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 Envelope Glycoprotein Clone B41dCT in complex with the broa...
| 全体 | 名称: HIV-1 Envelope Glycoprotein Clone B41dCT in complex with the broadly neutralizing antibody Fab PGT151 |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 Envelope Glycoprotein Clone B41dCT in complex with the broa...
| 超分子 | 名称: HIV-1 Envelope Glycoprotein Clone B41dCT in complex with the broadly neutralizing antibody Fab PGT151 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: HIV-1 Envelope Glycoprotein Clone B41_delCT
| 分子 | 名称: HIV-1 Envelope Glycoprotein Clone B41_delCT / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAAKKW VTVYYGVPVW KEATTT LFC ASDAKAYDTE VHNVWATHAC VPTDPNPQEI VLGNVTENFN MWKNNMVEQM HEDIISL WD QSLKPCVKLT PLCVTLNCNN VNTNNTNNST NATISDWEKM ETGEMKNCSF ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAAKKW VTVYYGVPVW KEATTT LFC ASDAKAYDTE VHNVWATHAC VPTDPNPQEI VLGNVTENFN MWKNNMVEQM HEDIISL WD QSLKPCVKLT PLCVTLNCNN VNTNNTNNST NATISDWEKM ETGEMKNCSF NVTTSIRD K IKKEYALFYK LDVVPLENKN NINNTNITNY RLINCNTSVI TQACPKVSFE PIPIHYCAP AGFAILKCNS KTFNGSGPCT NVSTVQCTHG IRPVVSTQLL LNGSLAEEEI VIRSENITDN AKTIIVQLN EAVEINCTRP NNNTRKSIHI GPGRAFYATG DIIGNIRQAH CNISKARWNE T LGQIVAKL EEQFPNKTII FNHSSGGDPE IVTHSFNCGG EFFYCNTTPL FNSTWNNTRT DD YPTGGEQ NITLQCRIKQ IINMWQGVGK AMYAPPIRGQ IRCSSNITGL LLTRDGGRDQ NGT ETFRPG GGNMRDNWRS ELYKYKVVKI EPLGIAPTAA KRRVVQREKR AVGLGAFILG FLGA AGSTM GAASMALTVQ ARLLLSGIVQ QQNNLLRAIE AQQHMLQLTV WGIKQLQARV LAVER YLRD QQLLGIWGCS GKIICTTNVP WNDSWSNKTI NEIWDNMTWM QWEKEIDNYT QHIYTL LEV SQIQQEKNEQ ELLELDKWDS LWNWFSISNW LWYIKIFIMI VGGLIGLRIV FTVLSII SR VRQGGGSGGG WSHPQFEK |
-分子 #2: Immunoglobulin G PGT151 Fab, Heavy chain
| 分子 | 名称: Immunoglobulin G PGT151 Fab, Heavy chain / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: RVQLVESGGG VVQPGKSVRL SCVVSDFPFS KYPMYWVRQA PGKGLEWVAA ISGDAWHVVY SNSVQGRFLV SRDNVKNTLY LEMNSLKIE DTAVYRCARM FQESGPPRLD RWSGRNYYYY SGMDVWGQGT TVTVSSASTK GPSVFPLAPS SKSTSGGTAA L GCLVKDYF ...文字列: RVQLVESGGG VVQPGKSVRL SCVVSDFPFS KYPMYWVRQA PGKGLEWVAA ISGDAWHVVY SNSVQGRFLV SRDNVKNTLY LEMNSLKIE DTAVYRCARM FQESGPPRLD RWSGRNYYYY SGMDVWGQGT TVTVSSASTK GPSVFPLAPS SKSTSGGTAA L GCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC DK |
-分子 #3: Immunoglobulin G PGT151, Light chain
| 分子 | 名称: Immunoglobulin G PGT151, Light chain / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 配列 | 文字列: DIVMTQTPLS LSVTPGQPAS ISCKSSESLR QSNGKTSLYW YRQKPGQSPQ LLVFEVSNRF SGVSDRFVGS GSGTDFTLRI SRVEAEDVG FYYCMQSKDF PLTFGGGTKV DLKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV ...文字列: DIVMTQTPLS LSVTPGQPAS ISCKSSESLR QSNGKTSLYW YRQKPGQSPQ LLVFEVSNRF SGVSDRFVGS GSGTDFTLRI SRVEAEDVG FYYCMQSKDF PLTFGGGTKV DLKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV TEQDSKDSTY SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE C |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.13 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2238 / 平均露光時間: 8.5 sec. / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)