+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8224 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a mutant type IV pilus machinery (delta N1 ring deletion in PilQ) in the closed state | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 50.0 Å | |||||||||
 データ登録者 データ登録者 | Gold VAM / Salzer R / Averhoff B | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2016 ジャーナル: J Biol Chem / 年: 2016タイトル: Topology and Structure/Function Correlation of Ring- and Gate-forming Domains in the Dynamic Secretin Complex of Thermus thermophilus. 著者: Ralf Salzer / Edoardo D'Imprima / Vicki A M Gold / Ilona Rose / Moritz Drechsler / Janet Vonck / Beate Averhoff /  要旨: Secretins are versatile outer membrane pores used by many bacteria to secrete proteins, toxins, or filamentous phages; extrude type IV pili (T4P); or take up DNA. Extrusion of T4P and natural ...Secretins are versatile outer membrane pores used by many bacteria to secrete proteins, toxins, or filamentous phages; extrude type IV pili (T4P); or take up DNA. Extrusion of T4P and natural transformation of DNA in the thermophilic bacterium Thermus thermophilus requires a unique secretin complex comprising six stacked rings, a membrane-embedded cone structure, and two gates that open and close a central channel. To investigate the role of distinct domains in ring and gate formation, we examined a set of deletion derivatives by cryomicroscopy techniques. Here we report that maintaining the N0 ring in the deletion derivatives led to stable PilQ complexes. Analyses of the variants unraveled that an N-terminal domain comprising a unique βββαβ fold is essential for the formation of gate 2. Furthermore, we identified four βαββα domains essential for the formation of the N2 to N5 rings. Mutant studies revealed that deletion of individual ring domains significantly reduces piliation. The N1, N2, N4, and N5 deletion mutants were significantly impaired in T4P-mediated twitching motility, whereas the motility of the N3 mutant was comparable with that of wild-type cells. This indicates that the deletion of the N3 ring leads to increased pilus dynamics, thereby compensating for the reduced number of pili of the N3 mutant. All mutants exhibit a wild-type natural transformation phenotype, leading to the conclusion that DNA uptake is independent of functional T4P. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8224.map.gz emd_8224.map.gz | 355 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8224-v30.xml emd-8224-v30.xml emd-8224.xml emd-8224.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8224.png emd_8224.png | 56.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8224 http://ftp.pdbj.org/pub/emdb/structures/EMD-8224 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8224 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8224 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8224_validation.pdf.gz emd_8224_validation.pdf.gz | 79 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8224_full_validation.pdf.gz emd_8224_full_validation.pdf.gz | 78.1 KB | 表示 | |
| XML形式データ |  emd_8224_validation.xml.gz emd_8224_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8224 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8224 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8224 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8224 | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8224.map.gz / 形式: CCP4 / 大きさ: 501 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8224.map.gz / 形式: CCP4 / 大きさ: 501 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 11.56 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
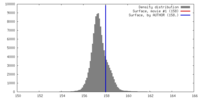
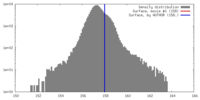
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Structure of a mutant type IV pilus machinery (delta N1 ring dele...
| 全体 | 名称: Structure of a mutant type IV pilus machinery (delta N1 ring deletion in PilQ) in the closed state from Thermus thermophilus |
|---|---|
| 要素 |
|
-超分子 #1: Structure of a mutant type IV pilus machinery (delta N1 ring dele...
| 超分子 | 名称: Structure of a mutant type IV pilus machinery (delta N1 ring deletion in PilQ) in the closed state from Thermus thermophilus タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus (バクテリア) / 株: HB27 Thermus thermophilus (バクテリア) / 株: HB27 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素:
| ||||||
|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 20 % / 装置: HOMEMADE PLUNGER 詳細: Cell solutions were mixed 1:1 (v/v) with 10 nm ProteinA-gold particles. 3 microlitres of sample were applied to grids and blotted on one side for ~5s before plunging. | ||||||
| 詳細 | Cellular suspension |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 3.0 e/Å2 詳細: Each image in every tilt series is a drift corrected sum of 3-5 frames |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 7.0 µm / 倍率(公称値): 10000 |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / ホルダー冷却材: NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C12 (12回回転対称) / アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 50.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF 詳細: Resolution estimate was calculated using a mask to exclude the membrane and peptidoglycan. C12 symmetry was applied to the masked central PilQ core by rotation of each subvolume by 30 degrees ...詳細: Resolution estimate was calculated using a mask to exclude the membrane and peptidoglycan. C12 symmetry was applied to the masked central PilQ core by rotation of each subvolume by 30 degrees (360 degrees/12) prior to alignment search. 使用したサブトモグラム数: 432 |
|---|---|
| 抽出 | トモグラム数: 8 / 使用した粒子像数: 36 / ソフトウェア - 名称:  IMOD (ver. 4.8.46) IMOD (ver. 4.8.46) |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)