+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8077 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MicroED structure of proteinase K at 1.75 A resolution | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peptidase K / serine-type endopeptidase activity / proteolysis / extracellular region / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Engyodontium album (菌類) Engyodontium album (菌類) | |||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / 解像度: 1.75 Å | |||||||||
 データ登録者 データ登録者 | Hattne J / Shi D | |||||||||
 引用 引用 |  ジャーナル: J Appl Crystallogr / 年: 2016 ジャーナル: J Appl Crystallogr / 年: 2016タイトル: Modeling truncated pixel values of faint reflections in MicroED images. 著者: Johan Hattne / Dan Shi / M Jason de la Cruz / Francis E Reyes / Tamir Gonen /  要旨: The weak pixel counts surrounding the Bragg spots in a diffraction image are important for establishing a model of the background underneath the peak and estimating the reliability of the integrated ...The weak pixel counts surrounding the Bragg spots in a diffraction image are important for establishing a model of the background underneath the peak and estimating the reliability of the integrated intensities. Under certain circumstances, particularly with equipment not optimized for low-intensity measurements, these pixel values may be corrupted by corrections applied to the raw image. This can lead to truncation of low pixel counts, resulting in anomalies in the integrated Bragg intensities, such as systematically higher signal-to-noise ratios. A correction for this effect can be approximated by a three-parameter lognormal distribution fitted to the weakly positive-valued pixels at similar scattering angles. The procedure is validated by the improved refinement of an atomic model against structure factor amplitudes derived from corrected micro-electron diffraction (MicroED) images. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8077.map.gz emd_8077.map.gz | 3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8077-v30.xml emd-8077-v30.xml emd-8077.xml emd-8077.xml | 12.9 KB 12.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8077.png emd_8077.png | 206.5 KB | ||
| Filedesc metadata |  emd-8077.cif.gz emd-8077.cif.gz | 5.5 KB | ||
| Filedesc structureFactors |  emd_8077_sf.cif.gz emd_8077_sf.cif.gz | 2.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8077 http://ftp.pdbj.org/pub/emdb/structures/EMD-8077 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8077 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8077 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8077_validation.pdf.gz emd_8077_validation.pdf.gz | 635.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8077_full_validation.pdf.gz emd_8077_full_validation.pdf.gz | 635.5 KB | 表示 | |
| XML形式データ |  emd_8077_validation.xml.gz emd_8077_validation.xml.gz | 4.3 KB | 表示 | |
| CIF形式データ |  emd_8077_validation.cif.gz emd_8077_validation.cif.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8077 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8077 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8077 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8077 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5i9sMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8077.map.gz / 形式: CCP4 / 大きさ: 3.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8077.map.gz / 形式: CCP4 / 大きさ: 3.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.60105 Å / Y: 0.60105 Å / Z: 0.5739 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
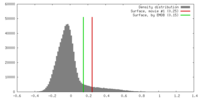
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 96 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Proteinase K
| 全体 | 名称: Proteinase K |
|---|---|
| 要素 |
|
-超分子 #1: Proteinase K
| 超分子 | 名称: Proteinase K / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Engyodontium album (菌類) Engyodontium album (菌類) |
| 分子量 | 理論値: 28.98 KDa |
-分子 #1: Proteinase K
| 分子 | 名称: Proteinase K / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: peptidase K |
|---|---|
| 由来(天然) | 生物種:  Engyodontium album (菌類) Engyodontium album (菌類) |
| 分子量 | 理論値: 28.958791 KDa |
| 配列 | 文字列: AAQTNAPWGL ARISSTSPGT STYYYDESAG QGSCVYVIDT GIEASHPEFE GRAQMVKTYY YSSRDGNGHG THCAGTVGSR TYGVAKKTQ LFGVKVLDDN GSGQYSTIIA GMDFVASDKN NRNCPKGVVA SLSLGGGYSS SVNSAAARLQ SSGVMVAVAA G NNNADARN ...文字列: AAQTNAPWGL ARISSTSPGT STYYYDESAG QGSCVYVIDT GIEASHPEFE GRAQMVKTYY YSSRDGNGHG THCAGTVGSR TYGVAKKTQ LFGVKVLDDN GSGQYSTIIA GMDFVASDKN NRNCPKGVVA SLSLGGGYSS SVNSAAARLQ SSGVMVAVAA G NNNADARN YSPASEPSVC TVGASDRYDR RSSFSNYGSV LDIFGPGTDI LSTWIGGSTR SISGTSMATP HVAGLAAYLM TL GKTTAAS ACRYIADTAN KGDLSNIPFG TVNLLAYNNY QA UniProtKB: Proteinase K |
-分子 #2: SULFATE ION
| 分子 | 名称: SULFATE ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: SO4 |
|---|---|
| 分子量 | 理論値: 96.063 Da |
| Chemical component information |  ChemComp-SO4: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 133 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線結晶学 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 25 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| ||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サイズ - 横: 2048 pixel / デジタル化 - サイズ - 縦: 2048 pixel / 撮影したグリッド数: 1 / 実像数: 184 / 回折像の数: 184 / 平均露光時間: 4.1 sec. / 平均電子線量: 0.004 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / カメラ長: 1200 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)