+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6595 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM of the C. elegans Ndc80 complex bound to microtubules | |||||||||
 マップデータ マップデータ | Helical reconstruction of the C. elegans Ndc80 complex bound to microtubules in the presence of the SKA1 complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Kinetochore protein complex | |||||||||
| 生物種 | unidentified (未定義) /  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.06 Å | |||||||||
 データ登録者 データ登録者 | Wilson-Kubalek EM / Cheeseman IM / Milligan RA | |||||||||
 引用 引用 |  ジャーナル: Mol Biol Cell / 年: 2016 ジャーナル: Mol Biol Cell / 年: 2016タイトル: Structural comparison of the Caenorhabditis elegans and human Ndc80 complexes bound to microtubules reveals distinct binding behavior. 著者: Elizabeth M Wilson-Kubalek / Iain M Cheeseman / Ronald A Milligan /  要旨: During cell division, kinetochores must remain tethered to the plus ends of dynamic microtubule polymers. However, the molecular basis for robust kinetochore-microtubule interactions remains poorly ...During cell division, kinetochores must remain tethered to the plus ends of dynamic microtubule polymers. However, the molecular basis for robust kinetochore-microtubule interactions remains poorly understood. The conserved four-subunit Ndc80 complex plays an essential and direct role in generating dynamic kinetochore-microtubule attachments. Here we compare the binding of theCaenorhabditis elegansand human Ndc80 complexes to microtubules at high resolution using cryo-electron microscopy reconstructions. Despite the conserved roles of the Ndc80 complex in diverse organisms, we find that the attachment mode of these complexes for microtubules is distinct. The human Ndc80 complex binds every tubulin monomer along the microtubule protofilament, whereas theC. elegansNdc80 complex binds more tightly to β-tubulin. In addition, theC. elegansNdc80 complex tilts more toward the adjacent protofilament. These structural differences in the Ndc80 complex between different species may play significant roles in the nature of kinetochore-microtubule interactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6595.map.gz emd_6595.map.gz | 15.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6595-v30.xml emd-6595-v30.xml emd-6595.xml emd-6595.xml | 9.6 KB 9.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6595.gif 400_6595.gif 80_6595.gif 80_6595.gif | 136.2 KB 8.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6595 http://ftp.pdbj.org/pub/emdb/structures/EMD-6595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6595 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6595_validation.pdf.gz emd_6595_validation.pdf.gz | 78.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6595_full_validation.pdf.gz emd_6595_full_validation.pdf.gz | 77.5 KB | 表示 | |
| XML形式データ |  emd_6595_validation.xml.gz emd_6595_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6595 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6595 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6595.map.gz / 形式: CCP4 / 大きさ: 16.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6595.map.gz / 形式: CCP4 / 大きさ: 16.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
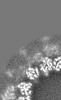
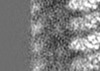
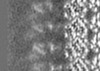
| 注釈 | Helical reconstruction of the C. elegans Ndc80 complex bound to microtubules in the presence of the SKA1 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
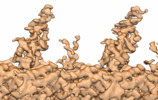
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
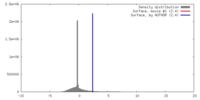
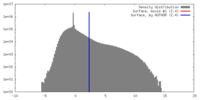
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : full-length C. elegans Ndc80 complex bound to microtubules
| 全体 | 名称: full-length C. elegans Ndc80 complex bound to microtubules |
|---|---|
| 要素 |
|
-超分子 #1000: full-length C. elegans Ndc80 complex bound to microtubules
| 超分子 | 名称: full-length C. elegans Ndc80 complex bound to microtubules タイプ: sample / ID: 1000 / 集合状態: helical / Number unique components: 2 |
|---|
-超分子 #1: microtubule
| 超分子 | 名称: microtubule / タイプ: organelle_or_cellular_component / ID: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
-分子 #1: Ndc80
| 分子 | 名称: Ndc80 / タイプ: protein_or_peptide / ID: 1 詳細: C. elegans Ndc80 complex was bound to the microtubule in the presence of the SKA1 complex 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 / 詳細: 80 mM PIPES, pH 6.8, 1 mM MgCl2, 1 mM EGTA |
|---|---|
| グリッド | 詳細: 400-mesh C-flat grids (Protochips, Inc) containing 2.0 micron holes separated by 2.0 micron spacing |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 120 K / 装置: HOMEMADE PLUNGER / 手法: Blotted back of grid for 3 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 日付 | 2015年9月23日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - サンプリング間隔: 1.31 µm / 実像数: 984 / 平均電子線量: 40 e/Å2 / 詳細: Each image is an average of 40 frames. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | EMAN2 with IHRSR adapted for microtubules with a dimer repeat and FREALIGN was used. |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 11.092 Å 想定した対称性 - らせんパラメータ - ΔΦ: 23.825 ° 想定した対称性 - らせんパラメータ - 軸対称性: C15 (15回回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 4.06 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, FREALIGN, IHRSR 詳細: The final map was calculated from two averaged data sets. |
| CTF補正 | 詳細: CTFIND v3 |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)