+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6462 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subvolumes of individual DNA minicircles showing various shapes of the minicircles | |||||||||
 マップデータ マップデータ | Subtomogram of a DNA minicircle | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA minicircle / cryo-ET / subtomogram / supercoiled DNA / DNA minicircle shapes | |||||||||
| 生物種 | unidentified (未定義) | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Irobalieva RN / Fogg JM / Catanese DJ / Sutthibutpong T / Chen M / Barker AK / Ludtke SJ / Harris SA / Schmid MF / Chiu W / Zechiedrich L | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2015 ジャーナル: Nat Commun / 年: 2015タイトル: Structural diversity of supercoiled DNA. 著者: Rossitza N Irobalieva / Jonathan M Fogg / Daniel J Catanese / Thana Sutthibutpong / Muyuan Chen / Anna K Barker / Steven J Ludtke / Sarah A Harris / Michael F Schmid / Wah Chiu / Lynn Zechiedrich /   要旨: By regulating access to the genetic code, DNA supercoiling strongly affects DNA metabolism. Despite its importance, however, much about supercoiled DNA (positively supercoiled DNA, in particular) ...By regulating access to the genetic code, DNA supercoiling strongly affects DNA metabolism. Despite its importance, however, much about supercoiled DNA (positively supercoiled DNA, in particular) remains unknown. Here we use electron cryo-tomography together with biochemical analyses to investigate structures of individual purified DNA minicircle topoisomers with defined degrees of supercoiling. Our results reveal that each topoisomer, negative or positive, adopts a unique and surprisingly wide distribution of three-dimensional conformations. Moreover, we uncover striking differences in how the topoisomers handle torsional stress. As negative supercoiling increases, bases are increasingly exposed. Beyond a sharp supercoiling threshold, we also detect exposed bases in positively supercoiled DNA. Molecular dynamics simulations independently confirm the conformational heterogeneity and provide atomistic insight into the flexibility of supercoiled DNA. Our integrated approach reveals the three-dimensional structures of DNA that are essential for its function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6462.map.gz emd_6462.map.gz | 14.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6462-v30.xml emd-6462-v30.xml emd-6462.xml emd-6462.xml | 9.5 KB 9.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6462.gif 400_6462.gif 80_6462.gif 80_6462.gif | 24.5 KB 2.4 KB | ||
| その他 |  emd_6462_additional_1.map.gz emd_6462_additional_1.map.gz emd_6462_additional_2.map.gz emd_6462_additional_2.map.gz emd_6462_additional_3.map.gz emd_6462_additional_3.map.gz emd_6462_additional_4.map.gz emd_6462_additional_4.map.gz | 6.1 MB 14.4 MB 6.1 MB 6.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6462 http://ftp.pdbj.org/pub/emdb/structures/EMD-6462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6462 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6462_validation.pdf.gz emd_6462_validation.pdf.gz | 78.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6462_full_validation.pdf.gz emd_6462_full_validation.pdf.gz | 77.3 KB | 表示 | |
| XML形式データ |  emd_6462_validation.xml.gz emd_6462_validation.xml.gz | 498 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6462 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6462 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6462.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6462.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram of a DNA minicircle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.52 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 6462 additional 1.map
| ファイル | emd_6462_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
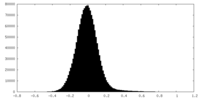
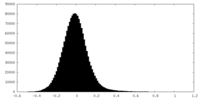
| 密度ヒストグラム |
-添付マップデータ: emd 6462 additional 2.map
| ファイル | emd_6462_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
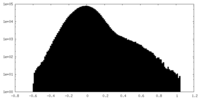
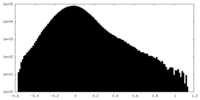
| 密度ヒストグラム |
-添付マップデータ: emd 6462 additional 3.map
| ファイル | emd_6462_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
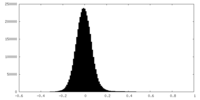
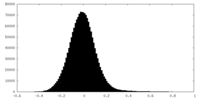
| 密度ヒストグラム |
-添付マップデータ: emd 6462 additional 4.map
| ファイル | emd_6462_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
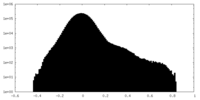
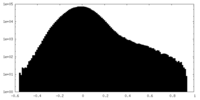
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Individual DNA minicircles of various shapes
| 全体 | 名称: Individual DNA minicircles of various shapes |
|---|---|
| 要素 |
|
-超分子 #1000: Individual DNA minicircles of various shapes
| 超分子 | 名称: Individual DNA minicircles of various shapes / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 220 KDa / 手法: average MW of DNA bases |
-分子 #1: Minicircle DNA
| 分子 | 名称: Minicircle DNA / タイプ: dna / ID: 1 / 分類: DNA / Structure: DOUBLE HELIX / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 220 KDa |
| 配列 | 文字列: TTTATACTAA CTTGAGCGAA ACGGGAAGGG TTTTCACCGA TATCACCGAA ACGCGCGAGG CAGCTGTATG GCATGAAAGA GTTCTTCCCG GAAAACGCGG TGGAATATTT CGTTTCCTAC TACGACTACT ATCAGCCGGA AGCCTATGTA CCGAGTTCCG ACACTTTCAT ...文字列: TTTATACTAA CTTGAGCGAA ACGGGAAGGG TTTTCACCGA TATCACCGAA ACGCGCGAGG CAGCTGTATG GCATGAAAGA GTTCTTCCCG GAAAACGCGG TGGAATATTT CGTTTCCTAC TACGACTACT ATCAGCCGGA AGCCTATGTA CCGAGTTCCG ACACTTTCAT TGAGAAAGAT GCCTCAGCTC TGTTACAGGT CACTAATACC ATCTAAGTAG TTGATTCATA GTGACTGCAT ATGTTGTGTT TTACAGTATT ATGTAGTCTG TTTTTTATGC AAAATCTAAT TTAATATATT GATATTTATA TCATTTTACG TTTCTCGTTC AGCTTT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 8.3 / 詳細: 40 mM Tris-acetate, 10 mM CaCl2 |
| グリッド | 詳細: 200 mesh holey carbon grids, glow-discharged for 25 seconds |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 95 K / 装置: FEI VITROBOT MARK IV / 手法: 1 blot, 2 seconds |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column omega energy filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 25.0 eV |
| 日付 | 2011年4月19日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 100 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 7.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 25000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° / Tilt series - Axis1 - Angle increment: 3 ° |
- 画像解析
画像解析
| 最終 再構成 | 使用した粒子像数: 41 |
|---|
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)