+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The chimeric flagellar motor complex between MotA1B1 from Paenibacillus sp. TCA20 and MotAB from E.coli, state 1 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Flagellar / motility / Paenibacillus / MOTOR PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bacterial-type flagellum stator complex / bacterial-type flagellum-dependent swarming motility / bacterial-type flagellum / proton channel activity / bacterial-type flagellum-dependent cell motility / proton transmembrane transport / chemotaxis / protein transport / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Paenibacillus sp. TCA20 (バクテリア) / Paenibacillus sp. TCA20 (バクテリア) /  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Onoe S / Nishikino T / Kinoshita M / Kishikawa J / Kato T | ||||||||||||
| 資金援助 |  日本, 3件 日本, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Biomolecules / 年: 2025 ジャーナル: Biomolecules / 年: 2025タイトル: Cryo-EM Structure of the Flagellar Motor Complex from sp. TCA20. 著者: Sakura Onoe / Tatsuro Nishikino / Miki Kinoshita / Norihiro Takekawa / Tohru Minamino / Katsumi Imada / Keiichi Namba / Jun-Ichi Kishikawa / Takayuki Kato /  要旨: The bacterial flagellum, a complex nanomachine composed of numerous proteins, is utilized by bacteria for swimming in various environments and plays a crucial role in their survival and infection. ...The bacterial flagellum, a complex nanomachine composed of numerous proteins, is utilized by bacteria for swimming in various environments and plays a crucial role in their survival and infection. The flagellar motor is composed of a rotor and stator complexes, with each stator unit functioning as an ion channel that converts flow from outside of cell membrane into rotational motion. sp. TCA20 was discovered in a hot spring, and a structural analysis was conducted on the stator complex using cryo-electron microscopy to elucidate its function. Two of the three structures (Classes 1 and 3) were found to have structural properties typical for other stator complexes. In contrast, in Class 2 structures, the pentamer ring of the A subunits forms a C-shape, with lauryl maltose neopentyl glycol (LMNG) bound to the periplasmic side of the interface between the A and B subunits. This interface is conserved in all stator complexes, suggesting that hydrophobic ligands and lipids can bind to this interface, a feature that could potentially be utilized in the development of novel antibiotics aimed at regulating cell motility and infection. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_63392.map.gz emd_63392.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-63392-v30.xml emd-63392-v30.xml emd-63392.xml emd-63392.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_63392_fsc.xml emd_63392_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_63392.png emd_63392.png | 96.8 KB | ||
| Filedesc metadata |  emd-63392.cif.gz emd-63392.cif.gz | 6.9 KB | ||
| その他 |  emd_63392_half_map_1.map.gz emd_63392_half_map_1.map.gz emd_63392_half_map_2.map.gz emd_63392_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-63392 http://ftp.pdbj.org/pub/emdb/structures/EMD-63392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-63392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-63392 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_63392_validation.pdf.gz emd_63392_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_63392_full_validation.pdf.gz emd_63392_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_63392_validation.xml.gz emd_63392_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_63392_validation.cif.gz emd_63392_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-63392 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-63392 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-63392 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-63392 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_63392.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_63392.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_63392_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
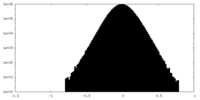
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_63392_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
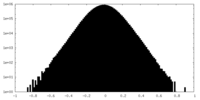
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The chimeric flagellar motor complex between MotA1B1 from Paeniba...
| 全体 | 名称: The chimeric flagellar motor complex between MotA1B1 from Paenibacillus sp. TCA20 and MotAB from E.coli |
|---|---|
| 要素 |
|
-超分子 #1: The chimeric flagellar motor complex between MotA1B1 from Paeniba...
| 超分子 | 名称: The chimeric flagellar motor complex between MotA1B1 from Paenibacillus sp. TCA20 and MotAB from E.coli タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 分子量 | 理論値: 150 KDa |
-超分子 #2: MotA
| 超分子 | 名称: MotA / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Paenibacillus sp. TCA20 (バクテリア) Paenibacillus sp. TCA20 (バクテリア) |
-超分子 #3: MotB1-MotB
| 超分子 | 名称: MotB1-MotB / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Paenibacillus sp. TCA20 (バクテリア) Paenibacillus sp. TCA20 (バクテリア) |
-分子 #1: Flagellar motor protein MotA
| 分子 | 名称: Flagellar motor protein MotA / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Paenibacillus sp. TCA20 (バクテリア) Paenibacillus sp. TCA20 (バクテリア) |
| 分子量 | 理論値: 28.568139 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDIATLIGLI AGAVAIIGGF LWEGGQITGL FQGTAALIVF GGTIAAVLIS YPMHRIRTLP AGIKLAFKPN RSEVNEWLED IVEMSMVAR REGVLALEQK VLDHPNIFLR EGIQLVVDGT DQPIVRQIME LDIDAKEQEH DNYAKLFESA GSYAPTMGII G TVMGLIQV ...文字列: MDIATLIGLI AGAVAIIGGF LWEGGQITGL FQGTAALIVF GGTIAAVLIS YPMHRIRTLP AGIKLAFKPN RSEVNEWLED IVEMSMVAR REGVLALEQK VLDHPNIFLR EGIQLVVDGT DQPIVRQIME LDIDAKEQEH DNYAKLFESA GSYAPTMGII G TVMGLIQV LGHLTDPSQL GPSIAVAFIA TLYGVASANL IFLPIASKIR AKSAEEILVM EMILEGVLSV QNGDNALLVR KK LNTYITS QPTSLNPRKD VTHETAE UniProtKB: Flagellar motor protein MotA |
-分子 #2: MotB1,Motility protein B
| 分子 | 名称: MotB1,Motility protein B / タイプ: protein_or_peptide / ID: 2 詳細: 6xHis on C terminal is purification tag,6xHis on C terminal is purification tag コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 35.262496 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRQRNRRTRN VKSAHSSGSP HDRWMITYAD LITLLLIFFV MMYAMSRLDA SKYEEVTSSL QTTFQSSSGI LDGGNGVIDY PSGQNGNSS SEANQPGSSG TGSDMGQEAD GGPLTERESR LRKLRGDLDQ LIESDPKLRA LRPHLKIDLV QEGLRIQIID S QNRPMFRT ...文字列: MRQRNRRTRN VKSAHSSGSP HDRWMITYAD LITLLLIFFV MMYAMSRLDA SKYEEVTSSL QTTFQSSSGI LDGGNGVIDY PSGQNGNSS SEANQPGSSG TGSDMGQEAD GGPLTERESR LRKLRGDLDQ LIESDPKLRA LRPHLKIDLV QEGLRIQIID S QNRPMFRT GSADVEPYMR DILRAIAPVL NGIPNRISLS GHTDDFPYAS GEKGYSNWEL SADRANASRR ELMVGGLDSG KV LRVVGMA ATMRLSDRGP DDAVNRRISL LVLNKQAEQA ILHENAESQN EPVSALEKPE VAPQVSVPTM PSAEPRHHHH HH UniProtKB: Motility protein B |
-分子 #3: Lauryl Maltose Neopentyl Glycol
| 分子 | 名称: Lauryl Maltose Neopentyl Glycol / タイプ: ligand / ID: 3 / コピー数: 1 / 式: AV0 |
|---|---|
| 分子量 | 理論値: 1.005188 KDa |
| Chemical component information |  ChemComp-AV0: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: CEOS Cs corrector / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 3129 / 平均露光時間: 3.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 3.295 µm / 最小 デフォーカス(補正後): 1.705 µm / 倍率(補正後): 56818 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.073 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-9lu9: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)