+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6255 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain reconstruction of the Pex1/Pex6 complex in presence of ATP | |||||||||
 マップデータ マップデータ | Negative stain reconstruction of the Pex1/Pex6 complex in presence of 3 mM ATP | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Peroxisome / AAA+ ATPase / Pex1 / Pex6 | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / ATP hydrolysis activity => GO:0016887 / protein targeting to peroxisome / protein import into peroxisome matrix, receptor recycling / protein binding / protein import into peroxisome matrix / peroxisome organization / transporter complex / protein transporter activity / peroxisomal membrane ...: / ATP hydrolysis activity => GO:0016887 / protein targeting to peroxisome / protein import into peroxisome matrix, receptor recycling / protein binding / protein import into peroxisome matrix / peroxisome organization / transporter complex / protein transporter activity / peroxisomal membrane / ATPase complex / protein unfolding / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / peroxisome / protein heterodimerization activity / nucleotide binding / ATP hydrolysis activity / ATP binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
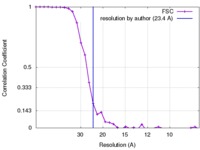
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 23.4 Å | |||||||||
 データ登録者 データ登録者 | Gardner BM / Chowdhury S / Lander GC / Martin A | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2015 ジャーナル: J Mol Biol / 年: 2015タイトル: The Pex1/Pex6 complex is a heterohexameric AAA+ motor with alternating and highly coordinated subunits. 著者: Brooke M Gardner / Saikat Chowdhury / Gabriel C Lander / Andreas Martin /  要旨: Pex1 and Pex6 are Type-2 AAA+ ATPases required for the de novo biogenesis of peroxisomes. Mutations in Pex1 and Pex6 account for the majority of the most severe forms of peroxisome biogenesis ...Pex1 and Pex6 are Type-2 AAA+ ATPases required for the de novo biogenesis of peroxisomes. Mutations in Pex1 and Pex6 account for the majority of the most severe forms of peroxisome biogenesis disorders in humans. Here, we show that the ATP-dependent complex of Pex1 and Pex6 from Saccharomyces cerevisiae is a heterohexamer with alternating subunits. Within the Pex1/Pex6 complex, only the D2 ATPase ring hydrolyzes ATP, while nucleotide binding in the D1 ring promotes complex assembly. ATP hydrolysis by Pex1 is highly coordinated with that of Pex6. Furthermore, Pex15, the membrane anchor required for Pex1/Pex6 recruitment to peroxisomes, inhibits the ATP-hydrolysis activity of Pex1/Pex6. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6255.map.gz emd_6255.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6255-v30.xml emd-6255-v30.xml emd-6255.xml emd-6255.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6255_fsc.xml emd_6255_fsc.xml | 3.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  400_6255.gif 400_6255.gif 80_6255.gif 80_6255.gif | 34.7 KB 4.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6255 http://ftp.pdbj.org/pub/emdb/structures/EMD-6255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6255 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6255_validation.pdf.gz emd_6255_validation.pdf.gz | 77.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6255_full_validation.pdf.gz emd_6255_full_validation.pdf.gz | 76.7 KB | 表示 | |
| XML形式データ |  emd_6255_validation.xml.gz emd_6255_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6255 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6255 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6255.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6255.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain reconstruction of the Pex1/Pex6 complex in presence of 3 mM ATP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pex1/Pex6 complex in presence of 3 mM ATP
| 全体 | 名称: Pex1/Pex6 complex in presence of 3 mM ATP |
|---|---|
| 要素 |
|
-超分子 #1000: Pex1/Pex6 complex in presence of 3 mM ATP
| 超分子 | 名称: Pex1/Pex6 complex in presence of 3 mM ATP / タイプ: sample / ID: 1000 詳細: 22 nM Pex1/Pex6 complex in presence of 3 mM ATP was used for negative stain electron microscopy. The sample was monodisperse. 集合状態: Heterohexamer containing three copies of Pex1 and three copies of Pex6 Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 707.076 KDa |
-分子 #1: Peroxisomal ATPase PEX1
| 分子 | 名称: Peroxisomal ATPase PEX1 / タイプ: protein_or_peptide / ID: 1 Name.synonym: Peroxin-1, Peroxisomal assembly protein 1, Peroxisome biogenesis protein PAS1 コピー数: 3 / 集合状態: Heterohexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 117 KDa / 理論値: 117 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Peroxisomal ATPase PEX1 GO: nucleotide binding, protein binding, ATP binding, cytoplasm, peroxisome, peroxisomal membrane, protein targeting to peroxisome, peroxisome organization, membrane, protein import into peroxisome ...GO: nucleotide binding, protein binding, ATP binding, cytoplasm, peroxisome, peroxisomal membrane, protein targeting to peroxisome, peroxisome organization, membrane, protein import into peroxisome matrix, receptor recycling, ATP hydrolysis activity, ATP hydrolysis activity => GO:0016887, protein heterodimerization activity InterPro: AAA+ ATPase domain, ATPase, AAA-type, core, ATPase, AAA-type, conserved site, CDC48 domain 2-like superfamily, P-loop containing nucleoside triphosphate hydrolase, Peroxisome biogenesis ...InterPro: AAA+ ATPase domain, ATPase, AAA-type, core, ATPase, AAA-type, conserved site, CDC48 domain 2-like superfamily, P-loop containing nucleoside triphosphate hydrolase, Peroxisome biogenesis factor 1, N-terminal, psi beta-barrel fold, Peroxisome biogenesis factor 1 |
-分子 #2: Peroxisomal ATPase PEX6
| 分子 | 名称: Peroxisomal ATPase PEX6 / タイプ: protein_or_peptide / ID: 2 Name.synonym: Peroxin-6 Peroxisomal assembly protein 8, Peroxisome biosynthesis protein PAS8 コピー数: 3 / 集合状態: Heterohexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 115 KDa / 理論値: 115 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Peroxisomal ATPase PEX6 GO: nucleotide binding, GO: 0001302, protein binding, ATP binding, cytoplasm, peroxisome, peroxisomal membrane, cytosol, peroxisome organization, membrane, protein import into peroxisome matrix, ...GO: nucleotide binding, GO: 0001302, protein binding, ATP binding, cytoplasm, peroxisome, peroxisomal membrane, cytosol, peroxisome organization, membrane, protein import into peroxisome matrix, receptor recycling, ATP hydrolysis activity, protein heterodimerization activity InterPro: AAA+ ATPase domain, ATPase, AAA-type, core, ATPase, AAA-type, conserved site, P-loop containing nucleoside triphosphate hydrolase |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.016 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 詳細: 60 mM HEPES, 50 mM NaCl, 50 mM KCl, 10 mM MgCl2, 0.5 mM EDTA, 1 mM TCEP, 3 mM ATP |
| 染色 | タイプ: NEGATIVE 詳細: 4 microliters of sample was applied to a freshly plasma-cleaned thin carbon surface that was pre-treated with 0.1% w/v poly-L-lysine hydrobromide. After removing excess protein, negative ...詳細: 4 microliters of sample was applied to a freshly plasma-cleaned thin carbon surface that was pre-treated with 0.1% w/v poly-L-lysine hydrobromide. After removing excess protein, negative staining was performed with 2% w/v uranyl formate solution. |
| グリッド | 詳細: 400 mesh Cu-Rh Maxtaform grid with a thin continuous carbon film on top |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 温度 | 最低: 294 K / 最高: 297 K / 平均: 295 K |
| アライメント法 | Legacy - 非点収差: Objective astigmatism was corrected using a quadrupole stigmator at 52,000 times magnification. |
| 詳細 | Data were collected at room temperature. |
| 日付 | 2014年11月26日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 825 / 平均電子線量: 20 e/Å2 詳細: Data collected with the Leginon automated image acquisition software. |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 52000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.20 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダー: Room temperature side entry holder / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)