+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of CasLambda2-crRNA-target DNA ternary complex in the NTS-cleaving state | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-CAS12 / GENOME ENGINEERING / HYDROLASE-RNA-DNA COMPLEX / DNA BINDING PROTEIN | |||||||||
| 生物種 | unidentified (未定義) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.93 Å | |||||||||
 データ登録者 データ登録者 | Omura SN / Hirano H / Itoh Y / Nureki O | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2025 ジャーナル: Commun Biol / 年: 2025タイトル: Structural basis for target DNA cleavage and guide RNA processing by CRISPR-Casλ2. 著者: Satoshi N Omura / Lauren E Alfonse / Alexa Ornstein / Hayato Morinaga / Hisato Hirano / Yuzuru Itoh / Gabrielle Munoz / Anthony J Garrity / Gregory R Hoffman / Tia DiTommaso / Winston X Yan / ...著者: Satoshi N Omura / Lauren E Alfonse / Alexa Ornstein / Hayato Morinaga / Hisato Hirano / Yuzuru Itoh / Gabrielle Munoz / Anthony J Garrity / Gregory R Hoffman / Tia DiTommaso / Winston X Yan / David R Cheng / David A Scott / Zachary Maben / Osamu Nureki /   要旨: RNA-guided CRISPR-Cas nucleases are widely used as versatile genome-engineering tools. Among the diverse CRISPR-Cas effectors, CRISPR-Casλ-also referred to as Cas12n-is a recently identified ...RNA-guided CRISPR-Cas nucleases are widely used as versatile genome-engineering tools. Among the diverse CRISPR-Cas effectors, CRISPR-Casλ-also referred to as Cas12n-is a recently identified miniature type V nuclease encoded in phage genomes. Given its demonstrated nuclease activity in both mammalian and plant cells, Casλ has emerged as a promising candidate for genome-editing applications. However, the precise molecular mechanisms of Casλ family enzymes remain poorly understood. In this study, we report the identification and detailed biochemical and structural characterizations of CRISPR-Casλ2. The cryo-electron microscopy structures of Casλ2 in five different functional states unveiled the dynamic domain rearrangements during its activation. Our biochemical analyses indicated that Casλ2 processes its precursor crRNA to a mature crRNA using the RuvC active site through a unique ruler mechanism, in which Casλ2 defines the spacer length of the mature crRNA. Furthermore, structural comparisons of Casλ2 with Casλ1 and CasΦ highlighted the diversity and conservation of phage-encoded type V CRISPR-Cas enzymes. Collectively, our findings augment the mechanistic understanding of diverse CRISPR-Cas nucleases and establish a framework for rational engineering of the CRISPR-Casλ-based genome-editing platform. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_61040.map.gz emd_61040.map.gz | 915.1 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-61040-v30.xml emd-61040-v30.xml emd-61040.xml emd-61040.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_61040_fsc.xml emd_61040_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_61040.png emd_61040.png | 110 KB | ||
| マスクデータ |  emd_61040_msk_1.map emd_61040_msk_1.map | 6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-61040.cif.gz emd-61040.cif.gz | 7 KB | ||
| その他 |  emd_61040_half_map_1.map.gz emd_61040_half_map_1.map.gz emd_61040_half_map_2.map.gz emd_61040_half_map_2.map.gz | 5.5 MB 5.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-61040 http://ftp.pdbj.org/pub/emdb/structures/EMD-61040 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-61040 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-61040 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_61040_validation.pdf.gz emd_61040_validation.pdf.gz | 753.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_61040_full_validation.pdf.gz emd_61040_full_validation.pdf.gz | 753.2 KB | 表示 | |
| XML形式データ |  emd_61040_validation.xml.gz emd_61040_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_61040_validation.cif.gz emd_61040_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-61040 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-61040 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-61040 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-61040 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_61040.map.gz / 形式: CCP4 / 大きさ: 6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_61040.map.gz / 形式: CCP4 / 大きさ: 6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.245 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_61040_msk_1.map emd_61040_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_61040_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_61040_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CasLambda2-crRNA-target DNA ternary complex
| 全体 | 名称: CasLambda2-crRNA-target DNA ternary complex |
|---|---|
| 要素 |
|
-超分子 #1: CasLambda2-crRNA-target DNA ternary complex
| 超分子 | 名称: CasLambda2-crRNA-target DNA ternary complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
-分子 #1: Cas Lambda2
| 分子 | 名称: Cas Lambda2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 88.746492 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHGG MKKSIKFKVK GNCPITKDVI NEYKEYYNKC SDWIKNNLTS ITIGEMAKFL QETLGKDVAY ISMGLSDEWK DKPLYHLFT KKYHTNNADN LLYYYIKEKN LDGYKGNTLN IGNTFFRQFG YFKLVVSNYR TKIRTLNCEI KRKKIDADST S EDIEMQTM ...文字列: MGHHHHHHGG MKKSIKFKVK GNCPITKDVI NEYKEYYNKC SDWIKNNLTS ITIGEMAKFL QETLGKDVAY ISMGLSDEWK DKPLYHLFT KKYHTNNADN LLYYYIKEKN LDGYKGNTLN IGNTFFRQFG YFKLVVSNYR TKIRTLNCEI KRKKIDADST S EDIEMQTM YEIIKHNLNK KTDWDEFISY IENVENPNID NINRYKLLRK CFCENENMIK NKLELLSIEQ LKNFGGCIMK QH INSMTLI IQHFKIEEKE NSLGFILNLP LNKKQYQIEL WGNRQVNKGT KERDAFLNTY GENIVFIINN DELYVVFSYE YEL EKEEAN FVKTVGLDVN FKHAFFVTSE KDNCHLDGYI NLYKYLLEHD EFTNLLTNDE KKDYEELSKV VTFCPFENQL LFAR YNKMS KFCKKEQVLS KLLYALQKQL KDENRTKEYI YVSCVNKLRA KYVSYFILKE KYYEKQKEYD IEMGFVDDST ESKES MDKR RTEFPFRNTP VANELLSKLN NVQQDINGCL KNIINYIYKI FEQNGYKIVA LENLENSNFE KKQVLPTIKS LLKYHK LEN QNVNDIKASD KVKEYIENGY YELITNENNE IVDAKYTEKG AMKVKNANFF NLMMKSLHFA SVKDEFVLLS NNGKTQI AL VPSEFTSQMD STDHCLYMKK NDKGKLVKAD KKEVRTKQEK HINGLNADFN AANNIKYIVE NEVWREIFCT RPKKAEYN V PSLDTTKKGP SAILHMLKKI EAIKILETEK |
-分子 #2: RNA (58-MER)
| 分子 | 名称: RNA (58-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 18.692006 KDa |
| 配列 | 文字列: GGCUUGUUGU AUAUAUUCUU UUAUAGGUAU UAAACAACGG AGAUGAGGUG CGCGUGGC |
-分子 #3: DNA (40-MER)
| 分子 | 名称: DNA (40-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 12.591749 KDa |
| 配列 | 文字列: (DT)(DG)(DT)(DC)(DT)(DA)(DT)(DT)(DT)(DA) (DG)(DG)(DA)(DG)(DA)(DT)(DG)(DA)(DG)(DG) (DT)(DG)(DC)(DG)(DC)(DG)(DT)(DG)(GS) (SC)(AS)(SC)(SC)(AS)(PST)(SC)(AS)(PST)(AS) (PST) |
-分子 #4: DNA (40-MER)
| 分子 | 名称: DNA (40-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 12.418669 KDa |
| 配列 | 文字列: (AS)(PST)(AS)(PST)(GS)(AS)(PST)(GS)(GS)(PST) (GS)(SC)(DC)(DA)(DC)(DG)(DC)(DG)(DC) (DA)(DC)(DC)(DT)(DC)(DA)(DT)(DC)(DT)(DC) (DC)(DT)(DA)(DA)(DA)(DT)(DA)(DG)(DA)(DC) (DA) |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: GUANOSINE-5'-THIO-MONOPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-THIO-MONOPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: GS |
|---|---|
| 分子量 | 理論値: 363.287 Da |
| Chemical component information |  ChemComp-GS: |
-分子 #7: 2-DEOXY-CYTIDINE-5'-THIOPHOSPHORATE
| 分子 | 名称: 2-DEOXY-CYTIDINE-5'-THIOPHOSPHORATE / タイプ: ligand / ID: 7 / コピー数: 3 / 式: SC |
|---|---|
| 分子量 | 理論値: 323.263 Da |
| Chemical component information |  ChemComp-SC: |
-分子 #8: 2-DEOXY-ADENOSINE -5'-THIO-MONOPHOSPHATE
| 分子 | 名称: 2-DEOXY-ADENOSINE -5'-THIO-MONOPHOSPHATE / タイプ: ligand / ID: 8 / コピー数: 2 / 式: AS |
|---|---|
| 分子量 | 理論値: 347.287 Da |
| Chemical component information | 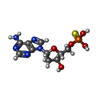 ChemComp-AS: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































