+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Orf virus scaffolding protein Orfv075 | |||||||||
 マップデータ マップデータ | 3D reconstruction of Orfv075 (sharpened) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Scaffold protein / Capsid protein / Poxvirus / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Poxvirus rifampicin-resistance / Poxvirus rifampicin resistance protein / response to antibiotic / membrane / 62 kDa protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Orf virus (strain NZ2) (ウイルス) Orf virus (strain NZ2) (ウイルス) | |||||||||
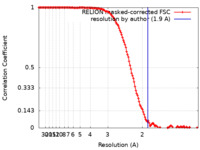
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.9 Å | |||||||||
 データ登録者 データ登録者 | Hyun J / Kim S / Ko S / Kim M / Jang Y | |||||||||
| 資金援助 |  韓国, 1件 韓国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biochem Biophys Res Commun / 年: 2024 ジャーナル: Biochem Biophys Res Commun / 年: 2024タイトル: Cryo-EM structure of orf virus scaffolding protein orfv075. 著者: Seungmi Kim / Sumin Ko / Minjae Kim / Yeontae Jang / Jaekyung Hyun /  要旨: Capsid-like poxvirus scaffold proteins self-assemble into semi-regular lattice that govern the formation of spherical immature virus particles. The scaffolding is a critical step in virus ...Capsid-like poxvirus scaffold proteins self-assemble into semi-regular lattice that govern the formation of spherical immature virus particles. The scaffolding is a critical step in virus morphogenesis as exemplified by the drug rifampicin that impairs the recruitment of scaffold onto the viral membrane in vaccinia virus (VACV). Here we report cryo-electron microscopy structure of scaffolding protein Orfv075 of orf virus (ORFV) that causes smallpox-like diseases in sheep, goats and occasionally humans via zoonotic infection. We demonstrate that the regions that are involved in intertrimeric interactions for scaffold assembly are largely conserved in comparison to its VACV orthologue protein D13 whose intermediate assembly structures have been previously characterized. By contrast, less conserved regions are located away from these interfaces, indicating both viruses share similar assembly mechanisms. We also show that the phenylalanine-rich binding site of rifampicin in D13 is conserved in Orfv075, and molecular docking simulation confirms similar binding modes. Our study provides structural basis of scaffolding protein as a target for anti-poxvirus treatment across wide range of poxvirus genera. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_60655.map.gz emd_60655.map.gz | 203.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-60655-v30.xml emd-60655-v30.xml emd-60655.xml emd-60655.xml | 22.8 KB 22.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_60655_fsc.xml emd_60655_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_60655.png emd_60655.png | 180.7 KB | ||
| マスクデータ |  emd_60655_msk_1.map emd_60655_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-60655.cif.gz emd-60655.cif.gz | 7 KB | ||
| その他 |  emd_60655_additional_1.map.gz emd_60655_additional_1.map.gz emd_60655_half_map_1.map.gz emd_60655_half_map_1.map.gz emd_60655_half_map_2.map.gz emd_60655_half_map_2.map.gz | 108.7 MB 200.7 MB 200.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-60655 http://ftp.pdbj.org/pub/emdb/structures/EMD-60655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60655 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_60655_validation.pdf.gz emd_60655_validation.pdf.gz | 1017.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_60655_full_validation.pdf.gz emd_60655_full_validation.pdf.gz | 1017.2 KB | 表示 | |
| XML形式データ |  emd_60655_validation.xml.gz emd_60655_validation.xml.gz | 20.6 KB | 表示 | |
| CIF形式データ |  emd_60655_validation.cif.gz emd_60655_validation.cif.gz | 26.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60655 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60655 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9ikcMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_60655.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_60655.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of Orfv075 (sharpened) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||
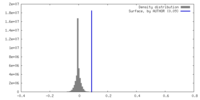
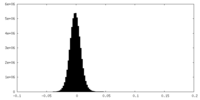
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_60655_msk_1.map emd_60655_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
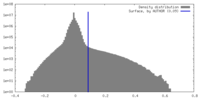
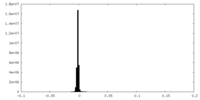
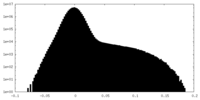
| 密度ヒストグラム |
-追加マップ: 3D reconstruction of Orfv075 (unsharpened)
| ファイル | emd_60655_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of Orfv075 (unsharpened) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
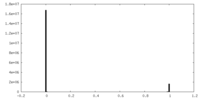
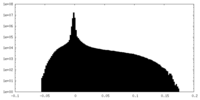
| 密度ヒストグラム |
-ハーフマップ: First halfmap of Orfv075 3D reconstruction
| ファイル | emd_60655_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | First halfmap of Orfv075 3D reconstruction | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
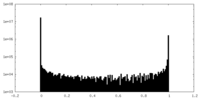
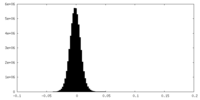
| 密度ヒストグラム |
-ハーフマップ: Second halfmap of Orfv075 3D reconstruction
| ファイル | emd_60655_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second halfmap of Orfv075 3D reconstruction | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
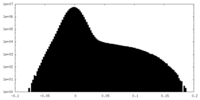
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Orfv075 trimer
| 全体 | 名称: Orfv075 trimer |
|---|---|
| 要素 |
|
-超分子 #1: Orfv075 trimer
| 超分子 | 名称: Orfv075 trimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Orf virus (strain NZ2) (ウイルス) Orf virus (strain NZ2) (ウイルス) |
| 分子量 | 理論値: 190 KDa |
-分子 #1: 62 kDa protein
| 分子 | 名称: 62 kDa protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Orf virus (strain NZ2) (ウイルス) Orf virus (strain NZ2) (ウイルス) |
| 分子量 | 理論値: 63.652172 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYYHHHHHH DYDIPTTENL YFQGAMNNSV VSLVGGDDAV RRASVFATDH RAPTVYMPQY ITTQGVVDTT SDAVTVTFEI RDKYISAMN NFVLSVDLPE IKGVGKMCYV PYIAYKLIRH VAVNSAADTI WETSGEELFD SCLDNERVME LSGFSRELND L STGSSPND ...文字列: MSYYHHHHHH DYDIPTTENL YFQGAMNNSV VSLVGGDDAV RRASVFATDH RAPTVYMPQY ITTQGVVDTT SDAVTVTFEI RDKYISAMN NFVLSVDLPE IKGVGKMCYV PYIAYKLIRH VAVNSAADTI WETSGEELFD SCLDNERVME LSGFSRELND L STGSSPND VIKEAACVHA YIKTPFDADK TFSTLKLSDS KVTVTVTLNP VACVMVYDET FDAAKLAKEF PYSMELSFIG YM VKNLCPR PAFIEMPRRR VEQINHTTAV ITDVHACTSL SVYMKPVLSD ANNRFISYPG FQQSEGDFVM AFVERLLEDM VIV SNCYPE GFPETAEIVE VPPSGVVSIQ DTDVFVRIDD VPVGMRVFLH TNILVFATRK NSVVYNMSKK FSAITGAYSR ATSR IRFTT AIHSVNIGDA SVPVGVWTCQ RNVYNGDNRS PEARAKDLFV ADPFLKGVDF KNKIDVIARM DVRFGNEVLY SENSA VSRV FGEILGKTPG VRTLQFNFTP STFFSPTALN SNVSRGKDKL AVRVTTAHME AHNPLMYVPR QMVVVCNEVY RLSYDA GIV AEKVTAQ UniProtKB: 62 kDa protein |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 150 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.13 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 93.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOCONTINUUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 10975 / 平均露光時間: 2.6 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 76923 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 1-547 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Structure of orthologue protein D13 (PDB 7VFD) was fitted into the density map of orfv075 using UCSF Chimera. The fitted structure was modified in Coot based on sequence alignment between D13 and Orfv075. The resulting Orfv075 model was refined using PHENIX. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-9ikc: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)