+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5856 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
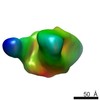
| タイトル | Negative stain reconstruction of HIV envelope glycoprotein BG505 SOSIP.664 in complex with CAP256-VRC26.09 Fab | |||||||||
 マップデータ マップデータ | Negative stain reconstruction of HIV-1 BG505 SOSIP.664 in complex with CAP256-VRC26.09 Fab | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV / envelope glycoprotein trimer / CAP256 / VRC.26 / V1/V2 directed antibody | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 28.0 Å | |||||||||
 データ登録者 データ登録者 | Ward AB / Kim HJ | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2014 ジャーナル: Nature / 年: 2014タイトル: Developmental pathway for potent V1V2-directed HIV-neutralizing antibodies. 著者: Nicole A Doria-Rose / Chaim A Schramm / Jason Gorman / Penny L Moore / Jinal N Bhiman / Brandon J DeKosky / Michael J Ernandes / Ivelin S Georgiev / Helen J Kim / Marie Pancera / Ryan P ...著者: Nicole A Doria-Rose / Chaim A Schramm / Jason Gorman / Penny L Moore / Jinal N Bhiman / Brandon J DeKosky / Michael J Ernandes / Ivelin S Georgiev / Helen J Kim / Marie Pancera / Ryan P Staupe / Han R Altae-Tran / Robert T Bailer / Ema T Crooks / Albert Cupo / Aliaksandr Druz / Nigel J Garrett / Kam H Hoi / Rui Kong / Mark K Louder / Nancy S Longo / Krisha McKee / Molati Nonyane / Sijy O'Dell / Ryan S Roark / Rebecca S Rudicell / Stephen D Schmidt / Daniel J Sheward / Cinque Soto / Constantinos Kurt Wibmer / Yongping Yang / Zhenhai Zhang / / James C Mullikin / James M Binley / Rogier W Sanders / Ian A Wilson / John P Moore / Andrew B Ward / George Georgiou / Carolyn Williamson / Salim S Abdool Karim / Lynn Morris / Peter D Kwong / Lawrence Shapiro / John R Mascola /    要旨: Antibodies capable of neutralizing HIV-1 often target variable regions 1 and 2 (V1V2) of the HIV-1 envelope, but the mechanism of their elicitation has been unclear. Here we define the developmental ...Antibodies capable of neutralizing HIV-1 often target variable regions 1 and 2 (V1V2) of the HIV-1 envelope, but the mechanism of their elicitation has been unclear. Here we define the developmental pathway by which such antibodies are generated and acquire the requisite molecular characteristics for neutralization. Twelve somatically related neutralizing antibodies (CAP256-VRC26.01-12) were isolated from donor CAP256 (from the Centre for the AIDS Programme of Research in South Africa (CAPRISA)); each antibody contained the protruding tyrosine-sulphated, anionic antigen-binding loop (complementarity-determining region (CDR) H3) characteristic of this category of antibodies. Their unmutated ancestor emerged between weeks 30-38 post-infection with a 35-residue CDR H3, and neutralized the virus that superinfected this individual 15 weeks after initial infection. Improved neutralization breadth and potency occurred by week 59 with modest affinity maturation, and was preceded by extensive diversification of the virus population. HIV-1 V1V2-directed neutralizing antibodies can thus develop relatively rapidly through initial selection of B cells with a long CDR H3, and limited subsequent somatic hypermutation. These data provide important insights relevant to HIV-1 vaccine development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5856.map.gz emd_5856.map.gz | 11.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5856-v30.xml emd-5856-v30.xml emd-5856.xml emd-5856.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_5856.gif 400_5856.gif 80_5856.gif 80_5856.gif | 22.6 KB 2.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5856 http://ftp.pdbj.org/pub/emdb/structures/EMD-5856 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5856 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5856 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5856_validation.pdf.gz emd_5856_validation.pdf.gz | 78.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5856_full_validation.pdf.gz emd_5856_full_validation.pdf.gz | 77.4 KB | 表示 | |
| XML形式データ |  emd_5856_validation.xml.gz emd_5856_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5856 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5856 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5856 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5856 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5856.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5856.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain reconstruction of HIV-1 BG505 SOSIP.664 in complex with CAP256-VRC26.09 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 BG505 SOSIP.664 in complex with CAP256-VRC26.09 Fab
| 全体 | 名称: HIV-1 BG505 SOSIP.664 in complex with CAP256-VRC26.09 Fab |
|---|---|
| 要素 |
|
-超分子 #1000: HIV-1 BG505 SOSIP.664 in complex with CAP256-VRC26.09 Fab
| 超分子 | 名称: HIV-1 BG505 SOSIP.664 in complex with CAP256-VRC26.09 Fab タイプ: sample / ID: 1000 / 詳細: Sample was monodisperse / 集合状態: One HIV trimer binds one Fab / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 410 KDa |
-分子 #1: envelope glycoprotein BG505 SOSIP.664
| 分子 | 名称: envelope glycoprotein BG505 SOSIP.664 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)別称: HIV-1 / 細胞中の位置: Membrane |
| 分子量 | 理論値: 360 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK 293S / 組換プラスミド: pPI4 Homo sapiens (ヒト) / 組換細胞: HEK 293S / 組換プラスミド: pPI4 |
-分子 #2: CAP256-VRC26.09 Fab
| 分子 | 名称: CAP256-VRC26.09 Fab / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 集合状態: heterodimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞: B cells Homo sapiens (ヒト) / 別称: Human / 細胞: B cells |
| 分子量 | 理論値: 50 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK 293F / 組換プラスミド: IgG1, kappa, lambda Homo sapiens (ヒト) / 組換細胞: HEK 293F / 組換プラスミド: IgG1, kappa, lambda |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.08 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50 mM Tris, 150 mM NaCl |
| 染色 | タイプ: NEGATIVE 詳細: Grids were adsorbed with protein, blotted, and stained with 2% uranyl formate for 20 seconds. |
| グリッド | 詳細: 400 Cu mesh grid with thin carbon support, glow discharged in natural atmosphere |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 温度 | 最低: 292 K / 最高: 294 K / 平均: 293 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 100,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 日付 | 2013年10月18日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 174 / 平均電子線量: 25 e/Å2 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 52000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle max: 50 |
- 画像解析
画像解析
| 詳細 | The particles were picked using the automated DoG Picker and put into a stack using the Appion software package. Initial reference free 2D class averages were calculated using particles binned by 2 via Xmipp Clustering 2D Alignment and sorted into classes. A template stack of 44 class averages was used to generate an ab initio model, which was refined using EMAN. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 28.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN, EMAN2, Xmipp, IMAGIC / 詳細: Map was calculated from a single dataset. / 使用した粒子像数: 6763 |
| 最終 2次元分類 | クラス数: 64 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















